淡水流域底栖动物eDNA宏条形码筛选研究论文
2024-11-06 11:56:43 来源: 作者:dingchenxi
摘要:利用eDNA宏条形码对淡水生态系统进行研究的过程中,通用基因的筛选很重要。本研究计算并比较了cox1、cob、12SrDNA和18SrDNA基因的进化速率和基因差异位点,结果显示cox1基因具有适中的进化速率和较高的种间差异,适合作为eDNA宏条形码的通用基因。
摘要:利用eDNA宏条形码对淡水生态系统进行研究的过程中,通用基因的筛选很重要。本研究计算并比较了cox1、cob、12SrDNA和18SrDNA基因的进化速率和基因差异位点,结果显示cox1基因具有适中的进化速率和较高的种间差异,适合作为eDNA宏条形码的通用基因。通过对比不同长度的cox1基因片段对结果的影响,发现300 bp左右的短片段在底栖动物物种多样性方面有较高的覆盖度。因此,本研究确定300 bp长度的cox1基因作为淡水底栖动物eDNA宏条形码的通用基因片段,以提高物种多样性评估的覆盖度,为淡水生态系统的生物多样性研究和生态修复提供技术服务。
关键词:eDNA宏条形码;底栖动物;线粒体cox1
近年来,国家高度重视流域水生态环境保护,对淡水流域底栖动物多样性调查是流域水生态保护的前提。eDNA宏条形码因其快捷、准确的特点成为淡水流域底栖动物多样性调查及水生态健康评价的研究热点[1-3]。目前在后生动物的DNA条形码研究中,普遍选择使用线粒体cox1等基因[4-6]。但淡水底栖动物不仅包含大量后生动物,也包括一些较为原始的动物类群,如多孔动物门等。针对不同类群,是否需要确定其他基因片段,如cob、12SrDNA、18SrDNA等基因作为DNA条形码通用基因,需要进一步研究。同时,这些基因的全长大部分都超过了1 000 bp。即使是常用的cox1基因经典引物LCO1490/HCO2198,所扩增出的序列长度也在658bp左右[7]。但在实际的水生环境中,生物体的DNA通常会受到紫外线、微生物分解等多种环境因素的影响而发生降解,形成较短的基因片段[8]。同时,eDNA宏条形码技术在进行高通量测序时,所使用的序列长度也相对较短。相较于传统引物,短基因片段引物PCR扩增效率更高[9]。如Leray等[10]确定了序列长度为313bp的cox1基因,用于研究天竺鲷科和金鳞鱼科珊瑚鱼的肠道内容物,与传统的658bp的cox1基因相比,这种段基因片段的扩增成功率更高。Vamos等[11]对GenBank中淡水大型无脊椎动物的基因序列进行了模拟测试,并设计了名为fwh1 set/fwh2 set的短基因片段引物(序列长度分别为178bp和205bp),在实际研究中应用表现良好。而且,引物的设计对于eDNA宏条形码的准确性有极大影响,如果引物与非目标生物碱基错配不成功,经常会出现PCR偏好性和假阳性、假阴性等问题,从而影响到生
物多样性的估算[12-14]。虽然短基因片段在eDNA宏条形码中更具优势,但并不是越短越好,太短的序列遗传信息不够,不足以鉴别物种。而且针对不同类群选择不同基因片段,片段的种类和长度没有统一的标准,这给eDNA宏条形码的大范围推广和应用造成了障碍[15-16]。因此,为了更有效地进行研究,我们需要针对底栖动物确定长度适中的基因片段及所对应的引物。
本研究计算了cox1、cob、12SrDNA、18SrDNA的基因进化速率,比较了它们的同义替换率Ks和非同义替换率Ka,非同义替换率和同义替换率的比率和基因差异位点分析,对cox1基因的长度进行了实验研究,最终确定300 bp左右的cox1基因可作为eDNA宏条形码的最佳通用基因。
1材料与方法
选取不同基因片段cox1、cob、12SrDNA和18SrDNA进行对比和计算。从GenBank中下载淡水底栖动物12个物种的不同基因片段,以蚓科的两个物种Lumbriculus variegatus和Stylodrilus heringianus作为参考,对照分析各个基因片段的进化参数。
选择山西省汾河流域的4个地点进行水样和泥样的采集,共采集到8个样本(A为水样,B为泥样)。对采集到的样本按照QIAGEN DNeasy Tissue kit使用手册的说明进行总DNA提取。并分别使用两种引物在ABI GeneAmp○R 9700型PCR仪上进行PCR扩增,最后利用Illumina PE250平台进行高通量测序,得到DNA宏条形码长度分别为650 bp及300bp左右进行OTU分析。
2结果与讨论
2.1进化速率计算
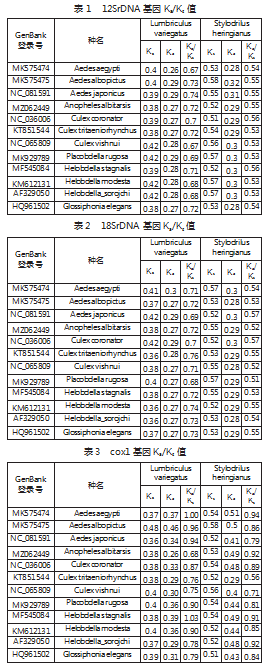
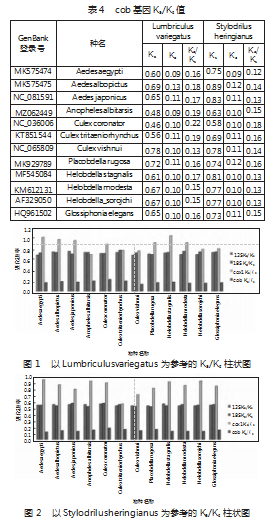
计算了12个物种的不同基因片段的同义替换率Ks和非同义替换率Ka,以及非同义替换率和同义替换率的比率,见表1—表4。同时,计算了各个基因片段的比率,取其平均值(见图1、图2)。从图和表中可以看出,18SrDNA进化速率最快,cox1进化速率居中,其他cob和12SrDNA的进化速率较慢。
2.2基因差异位点分析
通过对cox1、cob、12SrDNA、18SrDNA进行比对和计算,分析了种间基因差异位点和基因变异,如图3。从分析的结果可以看出,基因差异位点比例差异较大,12SrDNA、cob相对保守,差异位点比例分别为16%和13%,18SrDNA变异较快,差异位点比例为69%,cox1居中,为39%。Pi值与差异位点比例结果基本一致。

2.3 cox1基因长度的确定
对两种不同长度的基因片段进行注释分析,由表5得知,300 bp的短片段在目分类水平上的成功注释比例为94.95%,比长片段(650 bp)的67.93%更高。对注释成功的物种数量进行统计可知,650 bp的基因片段注释到3门5纲7目16科56属98种,300 bp的引物注释到4门6纲17目54科146属183种,300bp的基因片段注释到的底栖动物物种数量更多。
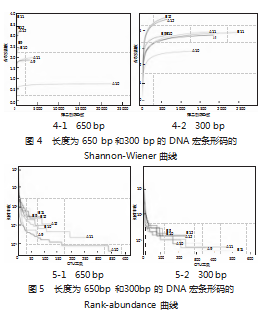
通过观察eDNA宏条形码技术所注释到的底栖动物Shannon-Wiener曲线(图4)显示,300 bp长度引物的Shannon-Wiener曲线更加趋于平缓,说明测序数据量足够,可以反映样本中绝大多数的底栖动物群落信息;同时,650bp的Shannon指数均值为2.52,300bp的Shannon指数均值为3.71,说明300bp的引物检测到的物种多样性更丰富。从Rank-abundance曲线(图5)来看,长片段A11、A10的OTU等级最高达到350左右,其余采样点的OTU均在150以下,而短片段A9、A11、B11、A12、B12的OTU等级均在200以上,且最大为520,因此短片段得到的底栖动物物种多样性更高。从曲线可以看出,300 bp引物的Rank-abundance曲线整体差异性也较小。
综上所述,短片段所得到的底栖动物物种多样性更高,生物差异性更小更稳定,更适宜作为DNA宏条形码通用基因片段。同时,因水中DNA序列在光照、高温等环境中,易降解为短片段,所以采用300 bp左右长度的cox1序列更适宜底栖动物DNA宏条形码的研究。
3结论
本研究综合各基因片段的Ka/Ks比率和种间基因差异位点和基因变异分析,可以看出,线粒体基因cob、12SrDNA和18SrDNA基因Ka/Ks<1,进化速率较慢,cox1基因的Ka/Ks接近1,进化速率居中,而且cox1基因除了用于系统发育分析之外,近年来被用于很多动物类群的物种鉴定、近缘种的区别等研究,可以作为淡水底栖动物DNA宏条形码研究的通用基因片段。对于环境DNA宏条形码而言,300 bp左右长度的短基因片段cox1更具优势,有着更大的测序深度,能覆盖大部分物种,因此在底栖动物DNA宏条形码的研究中,选择短基因片段所对应的引物进行PCR扩增和高通量测序的效果更好。
参考文献
[1]赵茜,潘福霞,李斌,等.基于环境DNA技术的黄河流域下游山区河流大型底栖动物群落多样性特征及其影响要素分析[J].湖泊科学,2024,36(2):523-536.
[2]王晨,陶孟,李爱民,等.基于环境DNA宏条形码技术的秦淮河生物多样性研究[J].生态学报,2022,42(2):611-624.
[3]邹艳婷,胡丹心,吴非霏,等.珠江流域常见鱼类及大型底栖动物DNA条形码空缺分析[J].生态学报,2024,44(4):1564-1574.
[4]廖馨,杨明柳.基于环境DNA的合浦榄根村退化红树林底栖动物多样性的研究[J].广西科学院学报,2021,37(3):288-298.
[5]CostaO F,DeWaardR J,BoutillierJ,et al.Biological identifications through DNA barcodes:the case of the Crustacea[J].Canadian Journal of Fisheries and Aquatic Sciences,2007,64(2):272-295.
[6]康子清,张银龙,吴永波,等.环境DNA宏条形码在生物多样性研究与监测中的应用[J].生物技术通报,2022,38(1):299-310.
[7]Hebert Paul D N,et al.Biological identifications through DNA bar-codes.Proceedings.Biological sciences,2003,270(1512):313-321.
[8]李萌,尉婷婷,史博洋,等.环境DNA技术在淡水底栖大型无脊椎动物多样性监测中的应用[J].生物多样性,2019,27(5):480-490.
[9]吕宏森,董智玲,闫卉果,等.长江上游鱼类环境DNA通用引物的选择与验证[J/OL].水产学报,1-17[2024-03-25].
[10]Matthieu,Y J Y,P C,et al.A new versatile primer set targeting a short fragment of the mitochondrial COI region for metabarcoding metazoan diversity:application for characterizing coral reef fish gut contents.[J].Frontiers in zoology,2013,10(1):34.
[11]Vamos,Ecaterina,Elbrecht,et al.Short COI markers for freshwater macroinvertebrate metabarcoding[J].Metabarcoding and Metage-nomics,2017(1):14625.
[12]李冉.环境DNA宏条形码技术在稻田节肢动物研究中的应用初探[D].杭州:浙江大学,2023.
[13]郭金,蒋娟,龙云川,等.基于eDNA宏基因组的草海湖泊硅藻群落及多样性分析[J/OL].环境科学研究,1-13[2024-03-25].
[14]薛棋文.基于eDNA宏条形码的太湖流域沉积物原生生物监测与生态健康评价研究[D].常州:常州大学,2021.
[15]王萌,金小伟,林晓龙,等.基于环境DNA-宏条形码技术的底栖动物监测及水质评价研究进展[J].生态学报,2021,41(18):7440-7453.
[16]林佳艳.基于环境DNA宏条形码技术的嘉陵江下游鱼类多样性研究[D].重庆:西南大学,2022.










