嗜热链球菌c-di-AMP合成酶的结构预测论文
2024-04-29 09:55:46 来源: 作者:liyuan
摘要:作为一种新兴第二信使分子,c-di-AMP具有全局调控胞内渗透压平衡、DNA 损伤和生物 膜形成等生理功能。本实验室在实验前期筛选出了一株高产胞外多糖的嗜热链球菌菌株 S-3,但未 对其进行 c-di-AMP信号通路研究。本研究在嗜热链球菌 S-3 全基因组中筛选出c-di-AMP 合成 酶(StDAC),对其二级结构进行预测,结果显示 StDAC 二级结构包含 42.98%α-螺旋、15.32%β-折叠 和 38.30%无规则卷曲。利用 SWISS-MODEL和 Modeller 对 StDAC 进行单
摘 要:作为一种新兴第二信使分子,c-di-AMP具有全局调控胞内渗透压平衡、DNA 损伤和生物 膜形成等生理功能。本实验室在实验前期筛选出了一株高产胞外多糖的嗜热链球菌菌株 S-3,但未 对其进行 c-di-AMP信号通路研究。本研究在嗜热链球菌 S-3 全基因组中筛选出c-di-AMP 合成 酶(StDAC),对其二级结构进行预测,结果显示 StDAC 二级结构包含 42.98%α-螺旋、15.32%β-折叠 和 38.30%无规则卷曲。利用 SWISS-MODEL和 Modeller 对 StDAC 进行单模板和多模板同源建模, 由 SWISS-MODEL 建立的模型通过了 Ramachandran、Errat 和 Verify-3D 评估,表明其结构较为合 理;而 Modeller 建立的模型仅通过了 Ramachandran 评估,总体结构评分较低。本研究预测的嗜热链 球菌 StDAC 三维模型为解析其结构和功能奠定了基础。
关键词:嗜热链球菌;c-di-AMP;生物信息学;二级结构;同源建模
嗜热链球菌(Streptococcus thermophilus)是一种 革兰氏阳性菌,也是一种公认的食品安全菌株,被广 泛应用于酸奶和奶酪等传统乳制品的生产过程中[1- 2]。为适应乳制品发酵过程中的酸性环境,感知细胞 外的信号并用信使分子来传递信号,对嗜热链球菌 的生存尤为重要。第二信使分子 c-di-AMP 广泛存 在于细菌、古生菌尤其是厚壁菌门中,与脂肪酸代 谢、钾离子稳态、渗透压调节、细胞毒力以及生物膜 形成等多种生理功能密切相关 [3-8]。为阐明 c-di- AMP 在嗜热链球菌中的调控机制,研究 c-di-AMP 合成酶的结构与功能非常有必要。细菌 c-di-AMP 合成酶 DAC 包含一个高度保守的 DAC/DisA_N 结 构域,可催化 ATP 或 ADP 合成 c-di-AMP。DAC 蛋 白活性与保守的 DGA(Asp-Gly-Ala)和 RHR(Arg- His-Arg)基序密切相关[9-12]。DAC 催化 c-di-AMP 合 成需要辅因子二价金属离子,用 DAC 三级结构可以预测可能的辅因子和关键结合位点[13]。
本实验室在实验前期在传统乳制品中筛选出了 一株高产胞外多糖的嗜热链球菌菌株 S-3,并对其 进行了全基因组测序[14]。结合 HMMER 预测和 NCBI 数据库比对在嗜热链球菌 S-3 基因组中预测出的 c-di-AMP 合成酶(命名为 StDAC),对其跨膜区和 二级结构进行预测 ,并通过 SWISS-MODEL 和 Modeller 进行三级结构模拟,为后续探究 StDAC 的 结构与功能奠定基础。
1 方法
1.1 二级结构预测
将 StDAC 氨基酸序列提交至蛋白质序列网络 分析 在线软 件(https://npsa-prabi.ibcp.fr/cgi-bin/ npsa_automat.plpage=/NPSA/npsa_server.html),选择 其中 9 个有代表性的二级结构预测方法:SOPM,HNN,MLRC,DPM,DSC,GOR Ⅰ , GOR Ⅲ , PHD , Predator 对 StDAC 二级结构进行预测 [15 -16] 。其中 HNN,MLRC,DSC,GORⅢ , PHD,Predator 为三态定 义法,“h ”代表α-螺旋、“e”代表 β-折叠、“c ”代表无 规则卷曲;SOPM,DPM 和 GOR Ⅰ为四态定义法,“h ” 代表α-螺旋、“e ”代表 β-折叠、“c”代表无规则卷 曲、“t ”代表 β-转角。为统一标准采用三态定义法对 二级结构进行比较分析。
1.2 SWISS-MODEL 单模板建模
将 StDAC 氨基酸序列提交至 SWISS-MODEL 蛋白分子建模服务器(https://swissmodel.expasy.org/) 进行单模板建模,返回 GMQE 值小于 1 且接近 1、 QMENA 值接近 0、氨基酸同源性大于 30%模板的 pdb 格式。建立的模型通过 Discovery studio 可视化 即可模拟 StDAC 三级结构[17] 。建模模板来自于美国 国 家 生 物 技 术 信 息 中 心 (National Center of Biotechnology Information,NCBI) 的 Structure 数据 库。
1.3 Modeller 多模板建模
将 StDAC 氨基酸序列提交至 Blast(https://blast. ncbi.nlm.nih.gov/Blast.cgi)、Phyre2(http://www.sbg. bio . ic . ac . uk / phyre2 / html / page . cgi? id = index)和 HHpred(https://toolkit.tuebingen.mpg.de/tools/hhpred) 在线数据库,搜索与 StDAC 具有同源性的模板。将 氨基酸同源性与软件评分较高的单模板依次通过脚 本 salign.py,align2d_mult.py 和 model_mult.py 进行 多模板建模,返回的 pdb 文件通过 Discovery studio 可视化即可模拟 StDAC 三级结构。
1.4 模型评估
将预测 的模 型提交 至 SAVES 在线 服务器 (https://saves.mbi.ucla.edu/), 根 据 提 示 分 别 对 Verify-3D,Errat 和 PROCHECK 中 Ramachandran 图 对模型的 3D 结构、晶体学和生理化学参数进行评 估。
2 结果分析与讨论
2.1 c-di-AMP 合成酶预测
利用 HMMER 预测和 NCBI 数据库比对在嗜热 链球菌 S-3 全基因组中发现的一个编码 DisA_N 结 构域的 c-di-AMP 合成酶基因(orf0696),将其编码的蛋白命名为StDAC。StDAC 分别与已报道的枯草 芽孢杆菌 c -di-AMP 合成酶 CdaA 和 DisA 拥有 47%和 33%的氨基酸序列相似性[10,18] 。用 PROTTER 在线分析软件对 StDAC 跨膜区进行预测,结果显示 StDAC 的第 7~52 个氨基酸为跨膜区,第 1~6 个氨 基酸和第 53~235 个氨基酸位于膜内侧。跨膜区预 测结果为 StDAC 异源表达提供了借鉴,可通过切除 第 7~52 个氨基酸提高可溶性蛋白含量。如图 1 所示。
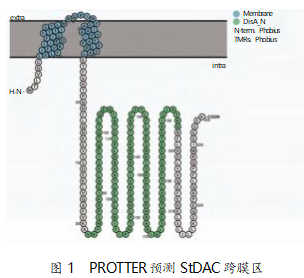
2.2 StDAC 二级结构预测
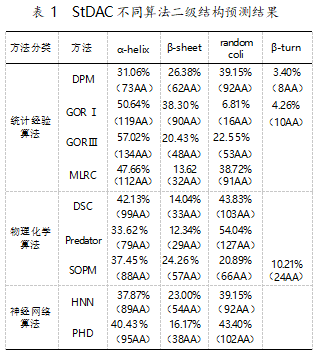
二级结构预测方法通常基于统计经验算法 (MLRC、DPM、GOR Ⅰ 、GOR Ⅲ )、物 理 化学 算 法 (SOPM、DSC、Predator)和神经网络算法(HNN、PHD), 这些方法预测的α-helix 的准确性高于 90%[19-20]。综 合 9 种预测方法得到的 StDAC 二级结构有 42.98% (共 101 个氨基酸)为 α-螺旋,15.32%(共 36 个氨基 酸)为 β-折叠,38.30%(共 90 个氨基酸)(图 2)为无 规则卷曲。用物理化学算法 DSC 和神经网络算法 PHD 预测的二级结构更接近综合预测结果。如表 1 所示。

2.3 StDAC 三级结构建模
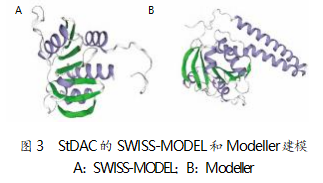
SWISS-MODEL 在线服务器选择用氨基酸同源 性最高和覆盖率最广的模板 6hvm 对 StDAC 进行单 模板建模(图 3A)。由于模板 6hvm 的晶体结构没有 跨膜区,因此 SWISS-MODEL 要对含 DisA_N 结构 域的 StDAC 从第 70~228 个氨基酸序列(共 158 个 氨基酸)进行三维建模。利用 Modeller 进行多模板建 模 ,用 NCBI、Phyre2 和 HHpred 在线数据库搜索StDAC 建模模板,以氨基酸同源性高、软件评分高和 氨基酸覆盖率广为标准,以 6gyw、6huw 和 d2fb5a1 为模板对 StDAC 进行多模板建模,如图 3B 所示。用 SWISS-MODEL 和 Modeller 两种方法建立的三级结 构都包含 5 个α-螺旋区域和 7 个 β-折叠区域,但 Modeller 建立的三级结构包括跨膜区域。
2.4 StDAC 三级结构评估
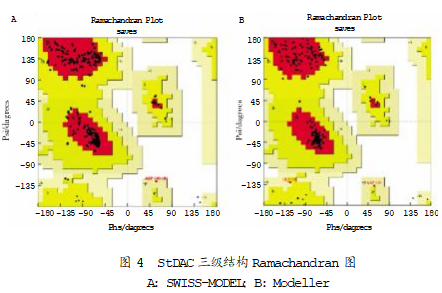
Ramachandran 图为最常见三级结构评估方法, 图中的红色区域为“最适区域”,黄色区域为“允许区域”,其他区域的红点均为构象不合理的氨基酸,合 理的三级结构须有 90%以上的氨基酸落在红色区 域 [21] 。用 SWISSMODEL 建立的 StDAC 三级结构有 92.0%的氨基酸落在最适区域,8.0%的氨基酸落在 允许区域。用 Modeller 建立的三级结构有 92.4%的 氨基酸落在最适区域,7.6%的氨基酸落在允许区 域 。 因 此 , 两 种 方 法 建 立 的 三 级 结 构 在 Ramachandran 图评估中都不存在氨基酸构象不合 理的情况。如图4 所示。
Errat 通过统计和分析 0.35 nm 半径内,不同类 型原子之间非键相互作用的数据,是通过晶体学方 法评估蛋白质模型,软件评分大于 85%的模型才具 有可靠性,图中的黄线和红线越多表示三级结构的 可靠性越差[22]。由 SWISSMODEL 建立的 StDAC 三级 结构 Errat 的评分为 98.98% ,模型通过评估。而 Modeller 建立的三级结构 Errat 的评分为 78.28%, 其中跨膜区和第 180~230 个氨基酸处出现了较多的黄线和红线,构象不合理导致模型评分低,如图 5 所示。

Verify-3D 是基于统计分析结果来评估蛋白三 维结构与氨基酸序列的相容性的,合理的三级结构 通常有 80%以上氨基酸的相容性评分高于 0.2[23]。由 SWISSMODEL建立的StDACVerify-3D评分为84.06%, 通过评估 。而 Modeller 建立 的 StDAC 三级结构 Verify-3D 的评分为 54.04%,没有通过评估,与 Errat 的评估结果相同,跨膜区和第 180~230 个氨基酸结 构不合理导致整体结构评分较低,如图 6 所示。
通过 Verify -3D、Errat 和 PROCHECK 中 的 Ramachandran图对建立的 StDAC 三级结构进行评 估,评估结果表明由 SWISS-MODEL 建立的 StDAC 三级结构通过了 Ramachandran、Errat 和 Verify-3D 的评估,蛋白结构较为合理;由 Modeller 建立的全长 StDAC 三级结构仅通过了 Ramachandran 评估,三级 结构评分低。因此,可通过 SWISS-MODEL 模拟的 三级结构对 StDAC 的结构与功能进行研究。
3 结论
基于嗜热链球菌 S-3 全基因组测序结果,结合 HMMER 预测和 NCBI 数据库 比对筛选出 c-di- AMP 合成酶 StDAC。利用统计经验算法、物理化学 算法和神经网络算法共 9 种方法预测 StDAC 的二 级 结 构 42.98%为 α -螺 旋、15.32%为 β -折 叠、 38.30%为无规则卷曲。分别通过 SWISS-MODEL 和 Modeller 模 拟 StDAC 三 级 结 构 ,其 中 SWISS - MODEL 单 模 板 建 模 的 StDAC 三 级 结 构 在 PROCHECK、Verify-3D 和 Errat 三个分析软件中评 分最高,为研究 c-di-AMP 合成酶 StDAC 的结构与 功能提供了可靠的模型,并为进一步阐明嗜热链球 菌中 c-di-AMP 的调控机制奠定了基础。
参 考 文 献
[1] 孔令慧, 赵林森, 夏永军, 等. 嗜热链球菌胞外多糖生物 合成的研究进展[J]. 食品安全质量检测学报, 2019, 10(2): 284-290.
[2] XU Z, GUO Q, ZHANG H, et al. Structural characterisation of EPS of Streptococcus thermophilus S-3 and its application in milk fermentation[J]. International Journal of Biological Macromolecules, 2021, 178(2): 263-269.
[3] GUNDLACH J, KRUGER L, HERZBERG C, et al. Sustained sensing in potassium homeostasis: cyclic di-AMP controls potassium uptake by KimA at the levels of expression and activity[J]. Journal of Biological Chemistry, 2019, 294(24): 9605-9614.
[4] CHENG X, ZHENG X, ZHOU X, et al. Regulation of oxidative response and extracellular polysaccharide synthesis by a diadenylate cyclase in Streptococcus mutans [J]. Environmental Microbiology, 2016, 18(3): 904-922.
[5] ZEDEN M S, KVIATKOVSKI I, SCHUSTER C F, et al. Identification of the main glutamine and glutamate transporters in Staphylococcus aureus and their impact on c-di-AMP production[J]. Molecular Microbiology, 2020, 113(6): 1085-1100.
[6] PENG X, MICHALEK S, WU H. Effects of diadenylate cyclase deficiency on synthesis of extracellular polysaccharide matrix of Streptococcus mutans revisit[J]. Environmental Microbiology, 2016, 18(11): 3612-3619.
[7] QUINTANA I M, GIBHARDT J, TURDIEV A, et al. The KupA and KupB Proteins of Lactococcus lactis IL1403 Are Novel c-di-AMP Receptor Proteins Responsible for Potassium Uptake[J]. Journal of Bacteriology, 2019, 201 (10): e00028-19.
[8] OPPENHEIMER-SHAANAN Y, WEXSELBLATT E, KATZHENDLER J, et al. c-di-AMP reports DNA integrity during sporulation in Bacillus subtilis[J]. European Molecular Biology Organization, 2011, 12(6): 594-601.
[9] HEIDEMANN J L, NEUMANN P, DICKMANNS A, et al. Crystal structures of the c-di-AMP-synthesizing enzyme CdaA[J]. Journal of Biological Chemistry, 2019, 294(27): 10463-10470.
[10] GANDARA C, ALONSO J C. DisA and c-di-AMP act at the intersection between DNA-damage response and stress homeostasis in exponentially growing Bacillus subtilis cells[J]. DNA Repair, 2015, 27(2015): 1-8.
[11] MEHNE F M, SCHRODER-TITTMANN K, EIJLANDER R T, et al. Control of the diadenylate cyclase CdaS in Bacillus subtilis: an autoinhibitory domain limits cyclic di-AMP production[J]. Journal of Biological Chemistry, 2014, 289(30): 21098-21107.
[12] BLOTZ C, TREFFON K, KAEVER V, et al.Identification of the components involved in cyclic di-AMP signaling in Mycoplasma pneumoniae[J]. Frontiers in Microbiology 2017, 8: 1328-1338.
[13] ROSENBERG J, DICKMANNS A, NEUMANN P, et al.Structural and biochemical analysis of the essential diadenylate cyclase CdaA from Listeria monocytogenes [J]. Journal of Biological Chemistry, 2015, 290 (10): 6596-6606.
[14] XIONG Z Q, KONG L H, MENG H L, et al. Comparison of gal lac operons in wild-type galactose-positive and -negative Streptococcus thermophilus by genomics and transcription analysis[J]. Journal of Industrial Microbiology and Biotechnology, 2019, 46(5): 751-758.
[15] 王艳春, 何东健, 王守志. 基于级联神经网络的蛋白质 二级结构预测[J]. 计算机工程, 2010, 36(4): 22-24.
[16] BURKHARD ROST C S. Prediction of protein secondary structure at better than 70% accuracy[J]. Journal of Molecular Biology, 1993, 232:584-599.
[17] SCHWEDE T, KOPP J, GUEX N, et al. SWISS-MODEL: An automated protein homology-modeling server[J]. Nucleic Acids Research, 2003, 31(13): 3381-3385.
[18] MEHNE F M, GUNKA K, EILERS H, et al. Cyclic di-AMP homeostasis in Bacillus subtilis: both lack and high level accumulation of the nucleotide are detrimental for cell growth[J]. Journal of Biological Chemistry, 2013, 288(3): 2004-2017.
[19] GEOURJON C D G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Comput Appl Biosci, 1995, 11(6): 681-684.
[20] DEL魪AGE G R B. An algorithm for protein secondary structure prediction based on class prediction[J]. Protein Engiueering, 1987, 1(4): 289-294.
[21] NEHA V KALMANKAR C R, P BALARAM. Sparsely populated residue conformations in protein structures: revisiting“experimental”Ramachandranmaps[J]. Proteins, 2014, 82(7): 1101-1112.
[22] C COLOVOS T O Y. Verification of protein structures Patterns of nonbonded atomic interactions[J]. Prorein Science, 1993, 2: 1511-1519.
[23] J U BOWIE R L, D EISENBERG. A method to identify protein sequences that fold into a known three-dimensional structure[J]. Science, 1991, 253(5016): 164-170.










