水生病毒宏基因组研究进展论文
2025-03-20 16:03:51 来源: 作者:xuling
摘要:病毒作为水生食物网的重要组成部分,是水域中含量最丰富的非细胞生命体。由于在病毒基因组中无法找到一个通用的标记基因,传统培养技术和聚合酶链式反应(PCR)方法在研究环境中病毒群落的多样性和动态性方面仍存在一定局限性。
摘要:病毒作为水生食物网的重要组成部分,是水域中含量最丰富的非细胞生命体。由于在病毒基因组中无法找到一个通用的标记基因,传统培养技术和聚合酶链式反应(PCR)方法在研究环境中病毒群落的多样性和动态性方面仍存在一定局限性。对此,病毒宏基因组学提供了一种更全面深入地研究环境中病毒的工具和方法,弥补了传统技术的局限。文章对近年来国内外关于水生病毒及病毒宏基因组的研究进展进行了综述。
关键词:水生病毒;病毒宏基因组;研究进展
水生病毒是水生生态系统中数量最丰富的生物体之一,在调控宿主生物群落、营养循环、基因转移、碳循环等方面具有至关重要的作用。传统水生病毒研究方法是通过培养分离技术得到纯种病毒。然而,水生病毒绝大多数不可培养,且缺乏类似16S rRNA和18S rRNA这样广泛存在且高度保守的标记基因进行鉴定。病毒宏基因组学的发展,为解决水生病毒研究过程中遇到的瓶颈带来了突破。文章整合了近年来有关水生病毒、病毒宏基因组的报道,概括总结了水生病毒宏基因组的研究进展。
1水生病毒概况
水生病毒在水域生态系统中扮演着重要的角色,既有利又有害。例如,噬藻体,是一类能专一感染蓝藻的噬菌体,在防治蓝藻水华方面潜力巨大,被公认为具有潜在控藻能力的生物因子。又如,水生呼肠孤病毒[1],可感染多种淡水或海水水生动物,极有可能造成水产养殖业的严重损失,对水产养殖业的危害不可忽视。因此,当前亟须对水生病毒展开更深入、综合、全面的研究。
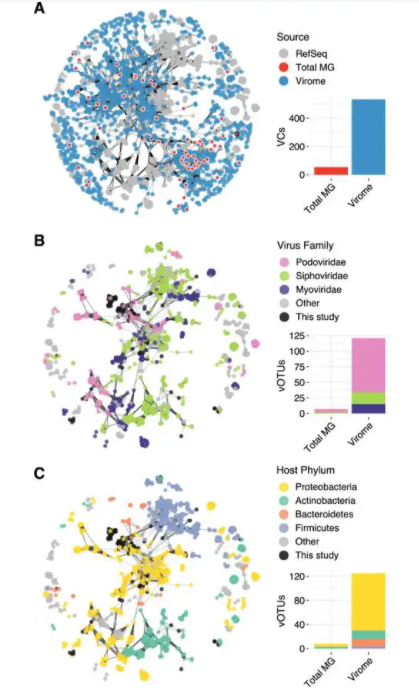
2病毒宏基因组学的概念
病毒宏基因组学是富集并提取来自环境或生物组织的病毒基因组,在对其测序后进行生物学信息分析的技术。该方法的发展得益于引物PCR和新一代测序技术的进步,不仅提高了病毒的研究效率,而且克服了以往研究中样本病毒浓度低和基因组易受干扰等问题。由于病毒宏基因组学的应用范围能够涵盖各种复杂多变的水域环境,其相关研究可以发现新的病毒种类、病毒群落的动态变化以及病毒与宿主之间的协同演化关系。
3病毒宏基因组学的研究方法
病毒宏基因组学的研究方法主要包括:样品预处理和病毒浓缩、病毒核酸的提取和扩增、病毒宏基因组学文库构建、高通量测序与数据分析。
3.1样品预处理和病毒浓缩
预处理样品可以最大程度保护DNA的完整性和质量,通过氯仿和DNase有效去除非病毒DNA,获得可靠的病毒宏基因组分析结果。该步骤一般获得的核酸含量很低,需对病毒进行浓缩。常用的浓缩方法有以下几种:切向流过滤,通过利用流体在滤膜表面的切向流动,以较低压力来分离和浓缩溶液中的颗粒,有效避免病毒衣壳受损或被破坏;氯化铁絮凝,通过添加氯化铁使病毒颗粒发生化学絮凝,并通过过滤器对絮凝的病毒颗粒进行分离和收集,是一种低成本、高效率的浓缩方法;离心超滤法,通过离心将样品中的病毒粒子聚集在离心管底部,并通过超滤膜将病毒粒子从其他成分中分离出来,是一种便捷的浓缩方法;氯化铯梯度,通过在密度梯度中离心,使不同密度的病毒粒子沉积到不同层次,从而实现对病毒的纯化。这些浓缩方法旨在提高样品中病毒粒子的纯度和浓度,以便进行后续的病毒宏基因组分析。
3.2病毒核酸的提取和扩增
病毒颗粒被纯化后,通常采用的核酸提取方法有两种:第一种是试剂盒法,不但方便提取高质量核酸,而且还具有较好的重复性;第二种是苯酚-氯仿提取法,虽然经济廉价,但提取率低,且存在苯酚被污染的问题,可能影响后续实验结果。核酸提取完毕后,需要进行污染检测,通过对小亚基rRNA基因进行PCR扩增,确定是否存在细胞DNA污染。同时,为了满足下一代测序的要求,通常需要对提取的核酸进行随机扩增。多重置换扩增和单引物序列非依赖性扩增是两种常用的扩增方法,可以将核酸扩增至足够浓度,以支持后续的测序分析。扩增完毕后还需进行电泳检测,若结果理想则可进行下一处理。
3.3病毒宏基因组学文库构建
高通量测序需要将样本的核酸转化为标准文库,以便在测序仪上进行装载和测序。对于双链DNA(dsDNA)病毒,可以直接构建宏基因组文库,而这种文库的构建可以通过在DNA两端添加适当的测序适配器来实现。对于RNA病毒,需要使用高效的反转录酶将RNA转录成单链cDNA,随后使用Klenow酶反应体系将其转换为双链cDNA,再构建文库。病毒宏基因组文库的克隆依赖型和克隆非依赖型是两种常见的构建方法。克隆依赖型文库是由克隆载体构建的基因文库,可以产生较大的连续序列或成对的终止读取序列,提供更好的注释信息,但生产成本高且耗时长,需要大量的实验操作和时间;克隆非依赖型文库则不需要克隆步骤,而是直接在核酸两端加上适当的接头,然后制成文库。相较而言,克隆非依赖型文库更适用于高通量测序技术,可以提供较长的读取序列,且误差较小。
3.4高通量测序与数据分析
最早的大规模海洋环境宏基因组测序工作是对马尾藻海表层水样的基因组文库进行鸟枪法测序,以揭示海洋微生物群落的多样性。随后,出现了以焦磷酸测序、合成测序、连接法测序为代表的第二代测序法,使病毒宏基因组学呈井喷式发展。单分子实时测序技术和纳米孔测序技术是第三代测序技术的代表,标志着新一轮的测序技术革命。在基因组学领域,高通量测序技术的诞生是一个具有里程碑意义的事件。其成本低廉、通量高、速度快,应用范围广,为许多研究者所喜爱。
高通量测序技术在病毒宏基因组学研究中产生了大量复杂数据,使数据分析成为一项关键挑战。通常情况下,针对病毒宏基因组学数据的分析流程包括以下几个主要步骤:数据质控、拼接组装、功能注释和基因预测。基因组装是将病毒基因组测序产生的reads通过序列拼接,且组装成完整的病毒基因组序列的过程。在病毒学领域,de Bruijn图算法常被应用于宏基因组装,组装结果会生成一系列Contigs序列。基因预测是对DNA序列中可能编码蛋白质的部分进行预测,即结构基因。目前,常用的基因预测方法包括基于统计学模型的方法和基于序列相似性及序列一致性的方法。使用同源比对软件(如BLAST等)将高通量测序生成的序列与本地数据库或公共数据库(如GenBank等)进行比对,可识别出可能的同源序列和相关信息。
4水生病毒宏基因组学
4.1海洋病毒宏基因组学
海洋中存在极为丰富且多样的病毒资源,而病毒宏基因组学的发展为海洋病毒的深度研究提供了有效的工具和方法。Yang等[2]从南极半岛附近的南斯科舍海岭采集水样,并对其进行病毒宏基因组分析,发现大多数病毒属于有尾病毒目,且球形棕囊藻噬菌体在调节病毒与宿主的相互作用方面可能扮演着重要的角色。Wu等[3]则对东海表层水中的病毒群落进行了深入分析,并通过荧光显微镜、透射电子显微镜和宏基因组学分析对其进行研究,发现了大量病毒样颗粒。这些研究证明了宏基因组分析在揭示海洋病毒多样性和功能方面的重要性。另外,Steward等[4]在200 m深的高压强海水中发现了病毒。这一发现带领人们将病毒研究视野拓展到了高压、高盐、高渗等极端环境。
4.2淡水病毒宏基因组学
病毒宏基因组学显著拓展了已知的病毒圈,但对淡水病毒的多样性研究较少。Skvortsov等[5]对来自爱尔兰内伊湖的病毒群落展开了深入的宏基因组分析,发现了比以往研究更丰富的病毒,特别是短尾病毒科。Okazaki等[6]对日本的琵琶湖(深淡水湖)的病毒进行了宏基因组探索,并发现放线菌病毒是淡水系统中最多样化、最普遍、最丰富的病毒群之一,且具有潜在的溶菌活性。
4.3污水病毒宏基因组学
噬菌体是污水的主要成分,Rosario等[7]通过对废水处理厂的污水样本进行病毒宏基因组学分析,揭示了污水中存在大量影响动植物和人类健康的致病性病毒。水体可通过该组学技术被快速检测,且得到的数据可作为判断其质量的直接指标,具有预防水生动物病毒性疾病的作用。
5总结
传统研究方法受限于对已知病毒的培养分离,难以挖掘更多新的水生病毒,而病毒宏基因组学方法结合新一代测序技术,可以不受培养限制地挖掘更多未知病毒,丰富对水域环境病毒多样性的认识。
病毒宏基因组学在病毒多样性、环境监测、生态系统功能研究等方面具有广泛的应用前景,虽然目前在复杂的生物信息学分析、数据的准确性和可靠性、样品处理的标准化等方面还存在技术瓶颈,但随着生物技术和信息技术的不断进步,相信这些问题会逐步得到解决。未来,病毒宏基因组学将与其他学科结合,形成更完善、全面的研究体系,为深入了解水域生物多样性和生态系统功能提供更多可能。
参考文献
[1]殷亮,王庆,曾伟伟,等.水生呼肠孤病毒基因组研究进展[J].动物医学进展,2013,34(1):80-85.
[2]YANG Q W,GAO C,JIANG Y,et al.Metagenomic characterization of the viral community of the south scotia ridge[J].Viruses,2019,11(2):95.
[3]WU S,ZHOU L,ZHOU Y F,et al.Diverse and unique viruses discovered in the surface water of the East China Sea[J].BMC Genomics,2020,21(1):441.
[4]Steward G F,Preston C M.Analysis of a viral metagenomic library from 200 m depth in Monterey Bay,California constructed by direct shotgun cloning[J].Virology Journal,2011,8(1):287.
[5]TIMOFEY S,COLIN L D,P J Q,et al.Metagenomic characterisation of the viral community of lough neagh,the largest freshwater lake in ireland[J].PloS One,2016,11(2):e0150361.
[6]YUSUKE O,YOSUKE N,TAKASHI Y,et al.Genome-resolved viral and cellular metagenomes revealed potential key virus-host interactions ina deep freshwater lake[J].Environmental Microbiology,2019,21(12):4740-4754.
[7]KARYNA R,CHRISTINA N,WEI Y L,et al.Metagenomic analysis of viruses in reclaimed water[J].Environmental Microbiology,2009,11(11):2806-2820.










