双歧杆菌蛋白表达系统的研究进展论文
2024-05-18 11:28:50 来源: 作者:liyuan
摘要:双歧杆菌是一种典型的益生菌,可被当作蛋白表达的生物安全级微生物底盘。重组双歧杆菌可以表达生物活性蛋白,具有广泛的食品应用价值和生物医学研究价值。文章综述了近年来对重组双歧杆菌蛋白表达应用的研究进展,以期为工程双歧杆菌的开发提供思路。
摘要:双歧杆菌是一种典型的益生菌,可被当作蛋白表达的生物安全级微生物底盘。重组双歧杆菌可以表达生物活性蛋白,具有广泛的食品应用价值和生物医学研究价值。文章综述了近年来对重组双歧杆菌蛋白表达应用的研究进展,以期为工程双歧杆菌的开发提供思路。
关键词:双歧杆菌;表达载体;表达体系;重组蛋白
随着人类基因组计划的完成,蛋白表达技术已逐渐渗透到生命科学研究的各个领域中,针对不同蛋白的生产需求,应该选用适当的表达系统及策略[1]。生物安全型的蛋白生产细胞工厂是近年来为满足食品及医药行业发展需求而形成的产物。益生菌是生物安全级微生物底盘,适合被用作蛋白生产宿主。双歧杆菌具有调节宿主肠道菌群和提高免疫力等作用,已被广泛应用于食品及医药领域[2-3]。而且,双歧杆菌在进行外源基因表达时,不易被改变益生活性,适合充当蛋白表达的宿主菌[4]。重组双歧杆菌的蛋白产物在改善菌株特性、辅助启动子筛选、示踪菌体体内定植,以及体内递送生物活性细胞因子等方面发挥着重要的作用。本文综述了近年来双歧杆菌表达重组蛋白的应用研究,以期为工程双歧杆菌的开发提供理论参考。
1双歧杆菌表达体系
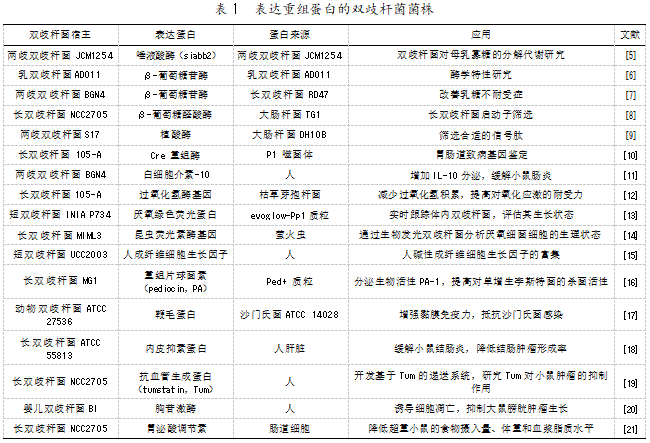
宿主菌及其表达载体是双歧杆菌蛋白表达体系的重要组成部分。双歧杆菌作为宿主菌,具有较强的蛋白分泌能力。同时,容易进行分子操作构建重组菌株也是双歧杆菌成为蛋白表达宿主的关键,包括成熟的宿主感受态细胞制备方法和高效转化的表达载体。近年来,应用于蛋白表达的双歧杆菌主要包括长双歧杆菌、短双歧杆菌、两歧双歧杆菌、动物双歧杆菌和婴儿双歧杆菌等(见表1)。其中,长双歧杆菌105-A和NCC2705、短双歧杆菌UCC2003及两歧双歧杆菌BGN4是被广泛应用的蛋白表达宿主,其表达的蛋白来源包括病毒、原核生物和真核生物。
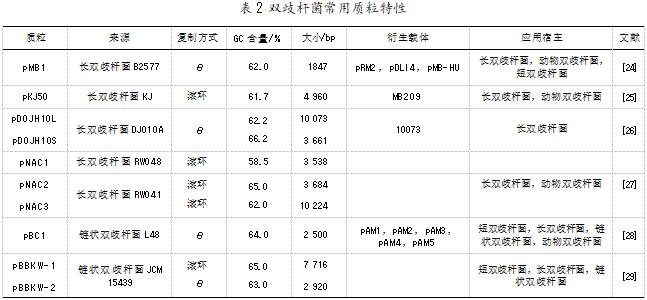
表达载体的遗传稳定性和高转化效率是蛋白表达的关键。双歧杆菌属包含的天然质粒较少,且由于菌株的特异性,导致其通用表达载体缺乏。部分常用的双歧杆菌载体的质粒特性溶氧仪如表2所示。目前,广泛应用的载体多源于长双歧杆菌,如pMB1、pKJ50、pDOJH10L和pNAC等。相比于其他双歧杆菌,长双歧杆菌基因组包含的隐形质粒较多,常被开发或改造成表达载体。例如,质粒pMB1来源于长双歧杆菌B2577,其衍生的质粒pRM2、pDLI4和pMB-HU被应用于动物双歧杆菌和短双歧杆菌。而来源于链状双歧杆菌L48的pBC1质粒也具有宿主广泛性,可应用于短双歧杆菌、长双歧杆菌和动物双歧杆菌等双歧杆菌的高效转化过程中。O’connell等[22]研究显示,由pBC1衍生的pAM系列质粒pAM5在短双歧杆菌UCC2003中的转化效率高达2×107 CFU/μg DNA。此外,研究还发现,质粒的复制方式对于构建穿梭载体的宿主范围、稳定性和拷贝数具有重要作用[23]。然而,目前尚无研究能够表明双歧杆菌中质粒的复制方式对转化效率有直接影响。
2重组双歧杆菌蛋白表达系统的应用
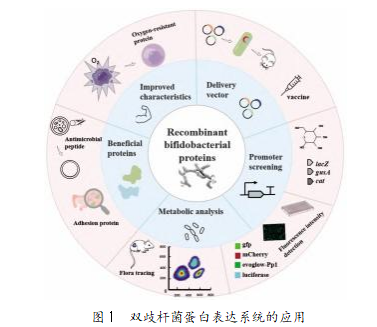
双歧杆菌蛋白表达系统集益生功能和活性蛋白表达的双重优点于一体,其主要应用如图1所示。例如,重组双歧杆菌可表达有益活性蛋白,增强菌株特性,其表达的碳水化合物酶和粘附蛋白可增强双歧杆菌在肠道中的定植能力;而异源抗氧化基因在双歧杆菌的表达可提高菌株的氧化应激能力。同时,重组双歧杆菌也可作为蛋白递送载体向体内输送生物活性因子。此外,双歧杆菌的蛋白表达产物还可作为报告基因用于启动子筛选和作为群体示踪的标志物。
2.1改善菌株特性
双歧杆菌基因组能编码广泛的酶库,分解和代谢各种碳水化合物底物,并且可以使双歧杆菌在胃肠道中保持活力和定植[30-31]。研究表明,人乳低聚糖可选择性促进双歧杆菌生长,如长双歧杆菌、婴儿双歧杆菌和短乳杆菌,并表达乳糖-N-四糖(lacto-N-tetraose,LNT)β-1,3-半乳糖苷酶分解人乳低聚糖,以促进其在婴幼儿肠道中定植[32]。双歧杆菌与肠上皮细胞(intestinal epithelial cells,IEC)的粘附是评估其在胃肠道中定植稳定性的重要因素。BopA是双歧杆菌特异性蛋白,可参与到对IEC的粘附的过程中。研究发现,表达BopA的双歧杆菌显示出了更强的肠道粘附力[33]。Gleinser等[5]分析了双歧杆菌表达重组细胞外唾液酸酶SiaBb2介导的肠道粘附机制,结果表明,重组蛋白SiaBb2可促进双歧杆菌在肠道细胞上的粘附,且有助于人乳寡糖和肠道糖复合物的同化。
通过引入外源抗氧化基因可显著提高双歧杆菌的抗逆性。研究发现,在长双歧杆菌NCC2705中表达嗜热链球菌SOD基因和植物乳杆菌血红素依赖性过氧化氢酶kat基因,可明显提高菌株氧化应激耐受性[34]。而且,O2可诱导长双歧杆菌JCM1255中异源TrxR-A hpC系统的表达,并通过降解H2O2保护菌体免受氧化应激[35]。Satoh等[12]在双歧杆菌中表达枯草芽孢杆菌katE基因,使其在H2O2环境下的存活率提高了100倍,通气条件下的存活率提高了105倍;其结果表明,双歧杆菌在应对氧化应激时表达异源基因比外源添加过氧化氢酶更有效。重组双歧杆菌的蛋白产物可改善菌株的特性,促进菌株生长,提高菌株的耐受力,有助于扩大基因工程双歧杆菌的应用范围,并完善对该菌属益生机制的研究。
2.2启动子筛选
启动子元件作为表达载体的重要组成部分,其强度直接影响着目标蛋白的表达水平。为筛选合适的启动子,通常需要建立不影响菌体生长的报告系统。易于检测的表型是报告系统的重要特征,包括发光报告系统和非发光报告系统。双歧杆菌中已成功表达了荧光蛋白、昆虫荧光素酶及细菌荧光素酶等发光报告基因。绿色荧光蛋白(Green fluorescent protein,GFP)通常在有氧条件下才能够表达,尽管在小部分双歧杆菌中可检测到荧光,但在不同双歧杆菌之间缺乏通用性[36]。恶臭假单胞菌evoglow-Pp1基因编码厌氧荧光蛋白,在应用于双歧杆菌中时可克服O2的限制。长双歧杆菌CECT4551以evoglow-Pp1为报告基因,通过筛选7种组成型启动子来追踪双歧杆菌,结果表明,含EF-P启动子的质粒在双歧杆菌中可观察到最高荧光强度[37]。非发光报告基因主要包括抗性基因和糖代谢相关基因。Kozakai等[38]构建了以氯霉素乙酰转移酶(chloramphenicolacetyltransferase,CAT)为报告基因的pKO403-TPCTcon质粒分析长双歧杆菌NCC2705的核心启动子。结果表明,双歧杆菌σ因子和启动子结构包含未知的极性结构域和核心序列盒,且不同于大肠杆菌的间隔区长度,表明细菌物种间存在不同的启动子结构。Sakanaka等[39]建立了基于双歧杆菌α-半乳糖苷酶的报告系统,并在长双歧杆菌和大肠杆菌中分析了不同双歧杆菌启动子的活性。
2.3群体示踪及代谢分析
报告系统可用于双歧杆菌菌群动力学分析[40]。含表达厌氧荧光蛋白的报告系统质粒可作为粪便中特定双歧杆菌的标记物,如携带pNZ:Tu-GFPana的长双歧杆菌CECT4551和短双歧杆菌INIAP734可直接通过绿色荧光细胞计数方法或荧光成像系统进行快速检测[41]。同时,基于荧光素酶的报告系统pLuxMC1可追踪双歧杆菌在体内外的生长情况,并证明短双歧杆菌UCC2003在小鼠体内是初次定植于盲肠,表明pLuxMC1具有充当双歧杆菌实时、非侵入性报告系统的潜力[42]。在非发光报告系统中,氯霉素抗性基因可作为pMGC-mCherry的标记物测定两歧双歧杆菌S17在胃肠道中的停留时间及其存活率[36]。但基于酶类的检测方法无法实时检测微生物,不适合用于评估体内特定双歧杆菌。荧光蛋白HaloTag,SNAP-tag和荧光激活吸收转移标签FAST已被视为厌氧丙酮丁醇梭菌和杨氏梭菌的有效报告基因,有助于扩展厌氧微生物的群体示踪及代谢分析工具,且有望应用于双歧杆菌的研究中[43]。
2.4蛋白递送载体
益生菌具有的耐酸耐胆盐特性及其在肠道中的粘附能力和定植能力,使其可在肠道内保持活性并释放目标物[44-45]。相较于其他益生菌,双歧杆菌的肠道定植能力具有普遍性,且适合作为递送蛋白质的载体[46],如辅助治疗低氧或厌氧环境下的肿瘤。Wang等[47]将包含pAM5的重组短双歧杆菌作为白介素-24的递送载体来观察不同时间小鼠头颈部肿瘤的治疗效果;结果表明,注射重组双歧杆菌可以抑制肿瘤生长并诱导肿瘤细胞凋亡,证明了双歧杆菌作为递送载体的有效性。Takata等[20]构建了含有内皮抑素蛋白的载体,并使其在pBV222-Endo长双歧杆菌ATCC55813中成功表达,以分析其对小鼠结肠癌的影响;结果表明,长期口服重组长双歧杆菌可显著降低肿瘤形成率、肿瘤数量和肿瘤大小。目前,双歧杆菌作为递送活载体的应用尚不成熟,对于双歧杆菌在肿瘤区域定植的准确性和有效释放能力,及其是否会引起机体的免疫反应等情况仍需进行深入研究[48-49]。
3总结与展望
双歧杆菌表达系统已经在食品和生物医药领域展现出了显著优势,但与其他有益菌群相比,双歧杆菌蛋白表达系统发展相对滞后,其菌株特异性是蛋白表达工具缺乏的关键因素。因此,开发通用的遗传操作方法和表达载体是双歧杆菌蛋白表达体系发展的重要途径。目前,可作为蛋白表达宿主菌的双歧杆菌菌株远远少于已公布基因组序列信息的双歧杆菌菌株。而利用比较基因组学深度挖掘未开发菌株的基因组信息,有助于扩展双歧杆菌蛋白表达宿主的范围。基因工程双歧杆菌可在短时间内获得目标蛋白产物,然而通常情况下,其表达的蛋白缺乏翻译后的修饰;同时,某些异源基因的持续表达也会增加宿主的生长负担,且表达产物常以包含体的形式出现,容易造成蛋白纯化困难的情况。因此,开发包含体蛋白表达和复性方法也有助于促进重组双歧杆菌表达系统的发展。此外,双歧杆菌作为工程菌尚未被直接应用于食品领域,未来的相关研究还需要对基因工程菌的安全性和适用性展开深入探讨和评估。随着双歧杆菌生物信息学和分子生物技术的发展,对双歧杆菌蛋白表达系统的研究将不断深入,并进一步扩展双歧杆菌表达重组蛋白的应用范围。
参考文献
[1]POROW I SKA D,WUJAK M,ROSZEK K,et al.Prokaryotic expression systems[J].Advances in Hygiene and Experimental Medicine,2013,67:119-129.
[2]VITHEEJONGJAROEN P,KANTHAWANG P,LOISON F,et al.Antioxidant activity of Bifidobacterium animalis MSMC83 and its application in set-style probiotic yoghurt[J].Food Bioscience,2021,43:101259.
[3]胡学智.益生元—双歧杆菌生长促进因子[J].工业微生物,2005,35(2):50-60.
[4]杨淼,赵笑笑,宋馨,等.双歧杆菌的分离培养和生理功能研究进展[J].工业微生物,2020,50(4):45-51.
[5]NISHIYAMA K,YAMAMOTO Y,SUGIYAMA M,et al.Bifidobacterium bifidum extracellular sialidase enhances adhesion to the mucosal surface and supports carbohydrate assimilation[J].MBio,2017,8(5):e00928-17.
[6]KIM J Y,WANG Y,PARK S J,et al.Cloning and expression ofβ-glucosidases from Bifidobacterium lactis AD011[J].Food Science and Biotechnology,2012,21:731-738.
[7]YOU H J,AHN H J,KIM J Y,et al.High expression ofβ-glucosidase in Bifidobacterium bifidum BGN4 and application in conversion of isoflavone glucosides during fermentation of soy milk[J].2015,25(4):469-78.
[8]WANG Y,KIM J Y,PARK M S,et al.Novel Bifidobacterium promoters selected through microarray analysis lead to constitutive high-level gene expression[J].Journal of Microbiology,2012,50:638-643.
[9]OSSWALD A,WESTERMANN C,SUN Z,et al.A phytase-based reporter system for identification of functional secretion signals in bifidobacteria[J].PLoS One,2015,10(6):e0128802.
[10]KOGUCHI H,ISHIGAMI N,SAKANAKA M,et al.Application of recombinase-based in vivo expression technology to Bifidobacterium longum subsp.longum for identification of genes induced in the gastrointestinal tract of mice[J].Microorganisms,2020,8(3):410.
[11]HONG N,KU S,YUK K,et al.Production of biologically active human interleukin-10 by Bifidobacterium bifidum BGN4[J].Microbial Cell Factories,2021,20(1):1-14.
[12]HE J,SAKAGUCHI K,SUZUKI T.Acquired tolerance to oxidative stress in Bifidobacterium longum 105-A via expression of a catalase gene[J].Applied and Environmental Microbiology,2012,78(8):2988-2990.
[13]HONG N,KU S,YUK K,et al.Production of biologically active human interleukin-10 by Bifidobacterium bifidum BGN4[J].Microbial Cell Factories,2021,20(1):1-14.
[14]LANDETE J M,PEIROTENÁ,RODRfGUEZ E,et al.Anaerobic green fluorescent protein as a marker of Bifidobacterium strains[J].International Journal of Food Microbiology,2014,175:6-13.
[15]GUGLIELMETTI S,SANTALA V,MANGAYIL R,et al.O2-requiring molecular reporters of gene expression for anaerobic microorganisms[J].Biosensors and Bioelectronics,2019,123:1-6.
[16]SHKOPOROV A N,EFIMOV B A,KHOKHLOVA E V,et al.Production of human basic fibroblast growth factor(FGF-2)in Bifidobacterium breve using a series of novel expression/secretion vectors[J].Biotechnology Letters,2008,30:1983-1988.
[17]MOON G S,PYUN Y R,PARK M S,et al.Secretion of recombinant pediocin PA-1 by Bifidobacterium longum,using the signal sequence for bifidobacterialα-amylase[J].Applied and Environmental Microbiology,2005,71(9):5630-5632.
[18]TAKATA T,SHIRAKAWA T,KAWASAKI Y,et al.Genetically engineered Bifidobacterium animalis expressing the Salmonella flagellin gene for the mucosal immunization in a mouse model[J].The Journal of Gene Medicine(A cross disciplinary journal for research on the science of gene transfer and its clinical applications),2006,8(11):1341-1346.
[19]BI Z,CUI E,YAO Y,et al.Recombinant Bifidobacterium longum carrying endostatin protein alleviates dextran sodium sulfate-induced colitis and colon cancer in rats[J].Frontiers in Microbiology,2022,13:927277.
[20]WEI C,XUN A Y,WEI X X,et al.Bifidobacteria expressing tumstatin protein for antitumor therapy in tumor-bearing mice[J].Technology in Cancer Research&Treatment,2016,15(3):498-508.
[21]YIN X,YU B,TANG Z,et al.Bifidobacterium infantis-mediated HSV-TK/GCV suicide gene therapy induces both extrinsic and intrinsic apoptosis in a rat model of bladder cancer[J].Cancer Gene Therapy,2013,20(2):77-81.
[22]O'CONNELL MOTHERWAY M,O'DRISCOLL J,FITZGERALD G F,et al.Overcoming the restriction barrier to plasmid transformation and targeted mutagenesis in Bifidobacterium breve UCC2003[J].Microbial Biotechnology,2009,2(3):321-332.
[23]肖路遥,石婷婷,王苏滢,等.副干酪乳酪杆菌ZY-1内源性质粒分析及其表达系统的构建与应用[J].生物工程学报,2023,39(3):1217-1231.
[24]ROSSI M,BRIGIDI P,Y RODRIGUEZ A G V,et al.Characterization of the plasmid pMB1 from Bifidobacterium longum and its use for shuttle vector construction[J].Research in Microbiology,1996,147(3):133-143.
[25]PARK M S,SHIN D W,LEE K H,et al.Sequence analysis of plasmid pKJ50 from Bifidobacterium longum[J].Microbiology,1999,145(3):585-592.
[26]LEE J H,O’SULLIVAN D J.Sequence analysis of two cryptic plasmids from Bifidobacterium longum DJO10A and construction of a shuttle cloning vector[J].Applied and Environmental Microbiology,2006,72(1):527-535.
[27]CORNEAU N,EMOND E,LAPOINTE G.Molecular characterization of three plasmids from Bifidobacterium longum[J].Plasmid,2004,51(2):87-100.
[28]Álvarez-Martín P,Fl 6rez A B,Mayo B.Screening for plasmids among human bifidobacteria species:sequencing and analysis of pBC1 from Bifidobacterium catenulatum L48[J].Plasmid,2007,57(2):165-174.
[29]TAKAHATA M,TOH H,NAKANO A,et al.Complete sequence analysis of two cryptic plasmids from Bifidobacterium kashiwanohense JCM 15439(type strain)isolated from healthy infant feces[J].Animal Science Journal,2014,85(2):158-163.
[30]O’CONNELL MOTHERWAY M,O’BRIEN F,O’DRISCOLL T,et al.Carbohydrate syntrophy enhances the establishment of Bifidobacterium breve UCC2003 in the neonatal gut[J].Scientific Reports,2018,8(1):10627.
[31]李刘若兰,张程程,刘秉书,等.不同假小链双歧杆菌对碳水化合物利用能力的比较研究[J].食品与发酵工业,49(14):94-102.
[32]ODAMAKI T,HORIGOME A,SUGAHARA H,et al.Comparative genomics revealed genetic diversity and species/strain-level differences in carbohydrate metabolism of three probiotic bifidobacterial species[J].International Journal of Genomics,2015,2015567809.
[33]GLEINSER M,GRIMM V,ZHURINA D,et al.Improved adhesive properties of recombinant bifidobacteria expressing the Bifidobacterium bifidum-specific lipoprotein BopA[J].Microbial Cell Factories,2012,11(1):1-14.
[34]XIAO M,XU P,ZHAO J,et al.Oxidative stress-related responses of Bifidobacterium longum subsp.longum BBMN68 at the proteomic level after exposure to oxygen[J].Microbiology,2011,157(6):1573-1588.
[35]ZUO F,YU R,FENG X,et al.Combination of heterogeneous catalase and superoxide dismutase protects Bifidobacterium longum strain NCC2705 from oxidative stress[J].Applied Microbiology and Biotechnology,2014,98:7523-7534.
[36]GRIMM V,GLEINSER M,NEU C,et al.Expression of fluorescent proteins in bifidobacteria for analysis of host-microbe interactions[J].Applied and Environmental Microbiology,2014,80(9):2842-2850.
[37]MONTENEGRO-RODRfGUEZ C,PEIROTEN A,SANCHEZ-JIMENEZ A,et al.Analysis of gene expression of bifidobacteria using as the reporter an anaerobic fluorescent protein[J].Biotechnology Letters,2015,37:1405-1413.
[38]KOZAKAI T,IZUMI A,HORIGOME A,et al.Structure of a core promoter in Bifidobacterium longum NCC2705[J].Journal of Bacteriology,2020,202(7):10.1128/jb.00540-19.
[39]SAKANAKA M,TAMAI S,HIRAYAMA Y,et al.Functional analysis of bifidobacterial promoters in Bifidobacterium longum and Escherichia coli using theα-galactosidase gene as a reporter[J].Journal of Bioscience and Bioengineering,2014,118(5):489-495.
[40]NINOMIYA K,YAMADA R,MATSUMOTO M,et al.Image analyzing method to evaluate in situ bioluminescence from an obligate anaerobe cultivated under various dissolved oxygen concentrations[J].Journal of Bioscience and Bioengineering,2013,115(2):196-199.
[41]LANDETE J M,PEIROTENÁ,RODR?GUEZ E,et al.Anaerobic green fluorescent protein as a marker of Bifidobacterium strains[J].International Journal of Food Microbiology,2014,175:6-13.
[42]CRONIN M,SLEATOR R D,HILL C,et al.Development of a luciferase-based reporter system to monitor Bifidobacterium breve UCC2003 persistence in mice[J].BMC Microbiology,2008,8:1-12.
[43]CHARUBIN K,STREETT H,PAPOUTSAKIS E T.Development of strong anaerobic fluorescent reporters for Clostridium acetobutylicum and Clostridium ljungdahlii using HaloTag and SNAP-tag proteins[J].Applied and Environmental Microbiology,2020,86(20):e01271-20.
[44]WOLFE W,XIANG Z,YU X,et al.The challenge of applications of probiotics in gastrointestinal diseases[J].
Advanced Gut&Microbiome Research,2023,2023:1-10.
[45]FEI Y,CHEN Z,HAN S,et al.Role of prebiotics in enhancing the function of next-generation probiotics in gut microbiota[J].Critical Reviews in Food Science and Nutrition,2023,63(8):1037-1054.
[46]马驰,杨涓,郑盛.肠道菌群与消化系统疾病的研究进展[J].现代医药卫生,2023,39(19):3349-3353,3359.
[47]WANG L,VULETIC I,DENG D,et al.Bifidobacterium breve as a delivery vector of IL-24 gene therapy for head and necksquamous cell carcinoma in vivo[J].Gene Therapy,2017,24(11):699-705.
[48]MAO N,CUBILLOS-RUIZ A,CAMERON D E,et al.Probiotic strains detect and suppress cholera in mice[J].Science Translational Medicine,2018,10(445):eaao2586.
[49]YAZAWA K,FUJIMORI M,AMANO J,et al.Bifidobacterium longum as a delivery system for cancer gene therapy:selective localization and growth in hypoxic tumors[J].Cancer Gene Therapy,2000,7(2):269-274.










