面向前列腺癌 Gleason 评分的深度学习方法研究现状综述论文
2024-04-26 14:06:58 来源: 作者:hemenglin
摘要:前列腺癌是一种常见的男性疾病, 严重危害着男性的身体健康。前列腺癌 Gleason 评分对于诊断前列腺癌及判断患者治疗后 的情况, 具有非常重要的意义
摘要:前列腺癌是一种常见的男性疾病, 严重危害着男性的身体健康。前列腺癌 Gleason 评分对于诊断前列腺癌及判断患者治疗后 的情况, 具有非常重要的意义。随着深度学习技术的快速发展, 涌现出了大量基于深度学习的前列腺癌 Gleason 评分方法。以不同 类型医学临床数据为研究对象, 可将现有面向前列腺癌 Gleason 评分的深度学习方法归纳为面向核磁共振图像、面向活检组织病理 图的前列腺癌 Gleason 评分方法。围绕上述研究对象, 对现有面向前列腺癌 Gleason 评分的深度学习方法进行分析和总结, 并对相 关技术的发展趋势进行展望。
关键词:前列腺癌; Gleason 评分;深度学习;核磁共振图像; 活检组织病理图
A Review of the Current State of Research on Deep Learning Methods for Gleason
Scoring of Prostate Cancer
Li Jia ,Cai Dingjian ,Huang Li ,Yang Min
(Faculty of Intelligent Manufacturing, Wuyi University, Jiangmen, Guangdong 529020. China)
Abstract: Prostate cancer is a common male disease that seriously endangers men ’s health. Gleason score for prostate cancer is very important for diagnosing prostate cancer and judging patients ’condition after treatment. With the rapid development of deep learning technology, a large number of deep learning-based Gleason scoring methods for prostate cancer have emerged. Taking different types of medical clinical data as the research objects, the existing deep learning methods for Gleason scoring of prostate cancer can be summarized into Gleason scoring methods for magnetic resonance images and Gleason scoring methods for biopsy histopathology images of prostate cancer. The existing deep learning methods for Gleason score of prostate cancer are analyzed and summarized, and the development trend of related technologies is prospected.
Key words: prostate cancer; Gleason score; deep learning; magnetic resonance images; biopsy histopathology images
引言
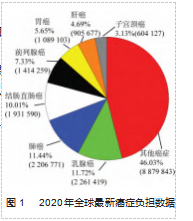
前列腺癌作为全球卫生保健的负担, 其发病率为 13.5%、死亡率为 6.7%, 是全球男性的第五大死亡原 因[1-2] ,也是世界上第四大常见癌症[3]。图 1 所示为前列 腺癌在 2020 年全球最新癌症负担数据中所占比例。老年男性是前列腺癌高发人 群, 据统计超过 80% 的 患者年龄在 65 岁以上[4], 仅 有 不 到 1% 的 前 列 腺 癌 患 者 年 龄 在 50 岁 以 下。前列腺癌前期诊断 非常重要, 若前期没有 及时发现并得到治疗 , 癌细胞会扩散到其他器 官中继续增殖, 进而危 及患者生命安全。前列腺特异性抗原(Prostate Specific Antigen , PSA) 是临床 上应用最广泛的前列腺癌筛选指标, 血清中 PSA 的检测 结果可以明显提高前列腺癌诊断的准确率。然而, PSA 及 PSA 相关指标均缺乏对前列腺癌特异性的表征, 导致 出现大量过度医疗[5] 的情况。针对上述问题,邢朋毅等[6] 利用前列腺癌泡癌中不同 Gleason 评分(Gleason Score, GS ) 的组织具有不同核磁共振图像纹理的特点, 采用随 机森林作为分类器实现对前列腺核磁共振图像的分类, 得到了高危组和低危组。 Fehr 等[7]结合表观扩散系数和 基于核磁共振图像的 T2 加权纹理特征, 提出了基于支持 向量机和自适应增强的自动分类方法, 实现了对不同 Gleason 评分的前列腺癌核磁共振图像分类, 评分包括 6 分(3 + 3)、 7 分(3 + 4)、 7 分(4 + 3)、大于 7 分, 其 中针对实验数据存在高度不平衡样本的问题, 采用了两 种不同的样本增强技术。
尽管上述基于传统机器学习的前列腺癌 Gleason 评分方法取得了一定的成效, 但是考虑到癌症患者的医学图 像中蕴含了复杂的与患病因素相关信息, 而深度学习能 够自动地挖掘数据中复杂的相关特征[8] ,并且在图像特 征识别和分析方面具有显著优势[9-10]。因此, 在现有前 列腺癌的诊断方法中, 深度学习作为传统机器学习技术 的替代方案, 已经被成功地应用在生物医学图像分析任 务中[11-14]。本文以核磁共振图像、活检组织病理图为例, 对现有面向前列腺癌 Gleason 评分的深度学习方法进行分 析和总结,并对相关技术的发展趋势进行展望。
1 Gleason 评分
Gleason 评分由 Gleason 分级确定。 Gleason 分级是一 种被广泛采用的前列腺癌组织学分级方法, 经典的 Glea ‐ son 分级被用来区分前列腺癌 5 种组织学生长模式, 按照 腺体的分化程度对前列腺癌活检图像分级, 分别是 Glea ‐ son1 级、 Gleason2 级、 Gleason3 级、 Gleason4 级、 Glea ‐ son5 级。其中 Gleason 1 级与预后良好更相关, 随着 Glea ‐ son 等级的升高, 患者的预后情况更趋近于不良。在对前 列腺活检图像评定 GS 时 Gleason1 级为 1 分, 表示最好的 细胞分化, 每增加一级增加一分, Gleason5 级为 5 分, 表示最坏的细胞分化。
在评定前列腺癌活检病理图 GS 时,对于同一个活检 切片可能存在多种不同的腺癌结构变异, 对其主要和次 要分化程度的组织分别进行评分, 将此两项评分相加得 到的总分作为整个活检切片的 GS, 积分为 2 、3 、4 分者 为高分化腺癌; 5 、6 、7 分者为中分化腺癌; 8 、9 、10 分者为低分化癌或者未分化癌。
Gleason 分级是由 Gleason 在 1966 年提出的, 后经过 1974 年、 1977 年两次修改。 Gleason 分级是评估前列腺 癌侵袭性最可靠的方法之一, 是预测前列腺癌预后情况 最有力的指标[15] 。2004 年, 世界卫生组织(WHO)批准 了 Gleason 分 级 , 它 也 被 列 入 AJCC/UICC 分 期 系 统 和 NCCN 指南。
2 面向前列腺癌 GS 评定的深度学习方法
尽管 GS 有助于前列腺癌的诊断和预后判断, 然而在 以往前列腺癌的诊断中, GS 通常是由病理学家在活检组 织病理图中确定的, 观察者之间存在显著的可变性, 因 此 GS 的评定具有主观性且难以执行[16]。最近的研究[17]建 议病理学家预测前列腺癌 GS 并量化不同 GS 肿瘤的百分 比, 这增加了病理学家的负担并加剧了主观性问题。基 于深度学习[18] 的自动分类方法能在该领域中一定程度上 减少人力的消耗及避免人为的主观失误。
2.1 核磁共振图像的前列腺癌 GS
核磁共振成像(Magnetic Resonance Imaging , MRI) 是临床上常用的医学影像检查手段。近年来, MRI 在诊 断前列腺癌中发挥着重要的作用, 多项研究已经确定前 列腺癌影像学特征与 GS[19-21]相关。特别是多参数核磁共振图像(multiparametric MRI ,mpMRI)方法更是提高了 MRI 评定前列腺癌 GS 的准确率, 可以更准确地检测出 有临床意义的前列腺癌, 即 GS≥ 7 和或肿瘤体积大于 0.5 cm3 的前列腺癌[22]。
然而目前 mpMRI 用于前列腺癌诊断仍受到定性或半 定量解释标准的限制, 导致评估病变侵袭性能力不佳。 基于上述问题, Cao 等[23] 提出了一种新的多分类卷积神 经网络 FocalNet, 用于检测前列腺癌并预测其 GS, 在训 练过程中, FocalNet 使用多种损失函数进行综合分析, 以充分利用来自 mpMRI 的信息。在临床上的实验结果表 明, FocalNet 的性能优于 3 种基于 CNN 的方法(Deeplab、 U-Net-Mult 、U-Net-Sing), 分 类 结 果 的 AUC 为 0.809. 然而, FocalNet 不能直接将病变实体识别为单个实体并 为每个实体评定 GS。这个问题能使用实例检测+分割的 方法解决[24]。此外, 已知 2 D 细胞图像神经网络在病变 检测任务中与 3 D 细胞图像神经网络相比表现不佳[25]。 因此, Oscar 等[26] 提出的基于深度学习全自动 3D Retina U-Net 是第一个利用适当的实例检测和分割网络, 同时 对前列腺中的病灶进行定位、分割和 GS 预测的,也是为 数不多的将 2 个截然不同的 mpMRI 数据集(ProstateX 数 据集和 IVO) 组合并输入到一个模型的作品之一 。Reti‐ na U-Net 架构将 Retina Net[27] 与 U-Net 相结合, 是专为处 理医学图像而设计的。在 Retina U-Net 中, 病变的检测 是独立于病变分割的(不同于其他类似的检测+分割架 构, 如 Mask R-CNN[24])。当 ProstateX 数据集作为测试集 时, 其分割得到的前列腺癌 GS 大于 2 的病灶的 AUC、敏 感性、特异性分别达到 0.96 、1.00 、0.79 ; 当测试数据为 IVO 数据集时, 其 AUC、敏感性、特异性分别达到 0.95、 1.00 、0.80 。Coen 等[28] 提出了一种以端到端方式同时检 测前列腺癌和对癌症组织评定 GS 的神经网络, 这比 ProstateX-2 挑战的分类目标任务更具有临床相关性; 提 出了一种在模型目标中编码 GS 的方法, 利用了类是有序 的这一事实。实验使用 ProstateX-2 挑战的数据集进行训 练, 使用前列腺 MRI 切片作为输入, 且使用带有编码 GS 的病变分割图作为输出。该模型在 ProstateX-2 挑战测试 集上得到的病灶加权 kappa 为 0.13±0.27. 结果表明该模 型优于标准的多分类和多标签序数回归。 Ahmad 等[29]提 出了一种新的基于深度熵特征(DEFs) 的放射组学方法 来预测 mpMRI 中前列腺癌的 GS。与传统的成像特征相 比, DEFs 是从卷积神经网络(CNN) 中学习的, 因此该 模型具有捕获图像更多信息特征的潜力。为了克服训练 数据有限的挑战, 所提出的方法使用了一种迁移学习策 略, 使用预训练的 CNN 提取成像特征, 然后将其总结为 一个小的 DEFs 集, 该 DEFs 集可以应用于预测前列腺癌 的 GS 。Luca 等[30]提出了一种基于多个卷积层的深度学习 框架, 旨在自动地将 GS 分配给所有 MRI。该深度学习框架具体由 4 个一维卷积层、 1 个 pooling 层、 1 个 flatten 层、 1 个 dropout 层和 1 个 dense 层共 8 个隐藏层组成。该方法 通过学习前列腺 MRI 中一组 71 个辐射特征预测得到 GS, 其预测准确率为 0.96~0.98.
2.2 活检组织病理图的前列腺癌 GS
第一个面向活检组织病理图前列腺癌 GS 评定的深度 学习方法是 Källén 等的研究[31] ,但类似于文献[32]中的想 法,其设计的是一种半自动的深度学习 GS 评定方法,将 预训练的 Overfeat[33]应用于提取病理图像特征。 Overfeat 是一个分为几个阶段的 22 层网络。第 1 阶段包括卷积层, 随后是分段线性函数, 后期阶段包括完全连接层和分类 层, 在一些阶段中, 还包括最大池化层。结果表明, 该 方 法 对 前 列 腺 活 检 组 织 病 理 图 评 定 GS 的 准 确 率 为 89.2% 。Bulten 等[34]使用 U-Net 架构开发了一种深度学习 方法来精确分割前列腺活检组织病理图中属于不同 GS 的 组织,并计算各个 GS 组织所占总百分比,使用这些百分 比直接确定整个腺体的 GS 。Lokhande 等[35]提出基于卷积 神经网络的 Carcino-Net, 用于将前列腺活检组织图分割 并分类为良性和 GS 分别为 3 、4 、5 的组织。 Carcino-Net 是一种改进型 FCN8s, 采用了 ResNet50 骨干网, 旨在提 高 FCN 的多类分割性能, 以在模型性能、计算资源和训 练图像数量之间进行更好的权衡。其首先在编码器部分 引入了一种改进: 使用 ResNet50 取代了参数量巨大的 VGG 来减少推理时间。由于残差块的引入, ResNet50 解 决了训练深度的问题, 从而降低了计算成本, 有助于避 免梯度消失问题。其次, 在解码器部分保留了 FCN8 的 EWS 层。解码器中的 EWS 层通过结合粗略和精细的语义 特征来帮助生成精确的分割边界。此外, Carcino-Net 还 在编码器的顶部添加了一个金字塔池化模块[36] ,而不是 平均池化层, 这使得 Carcino-Net 能够提取不同尺度的上 下文特征图。在 2019 MICCAI Gleason 挑战中, Carcino- Net 获得了第二名。 Kott 等[37]使用一种 18 层深的残差卷积 神经网络[38]根据 GS 将活检组织切片分类。其首先对活检 切片粗分类, 分类为良性或者恶性, 再对恶性的活检切 片根据 GS 细化分类。实验表明所提出的模型粗分类的准 确 率 为 91.5%, 细 分 类 的 准 确 率 为 85.4% 。 Kalapahar 等[39] 提出的基于残差块的 U-Net 架构的修改是自动深度 分割方法首次用于前列腺活检中不同 GS 的完全分类。该 方法根据完整的 GS 对前列腺癌变组织进行分割, 实验结 果表明该方法效果优于 FCN8 模型、标准的 UNet 以及 Segnet[40] , 在测试队列中达到了像素级的 Cohen ’s qua‐ dratic kappa 为 0.52 。Ryu 等[41] 提出了一种自动的基于深 度学习的前列腺癌 GS 评定方法,用于前列腺核心针活检 图的 GS 评定。所提出的方法包括两个步骤, 分别为 patch 级和图像级的 GS 评定。在第一步中, 活检图像被 分割成 704 像素×704 像素大小的 patch, 然后该深度学习方法根据 5 个类别对每个 patch 分类, 这 5 个类别分别为 非癌性、 GS3 、GS4 、GS5 以及前列腺导管内癌。在第二 步中,将每个 patch 的结果聚合到单个热力图中, 热力图 中每种 GS 区域的比例决定了活检图最终的 GS。该深度 学习方法由两个具有相同结构的基于 DeepLab v3+[42] 的人 工神经网络组成, 附加的非局部块[43]与 DeepLab v3+的编 码器、解码器之间的连接放置在一起。对 1 133 例前列腺 核心针活检样本进行训练, 并在 700 例病例中进行验证。 将诊断结果与来自 3 位经过认证的病理学家的诊断结果 进行比较。结果显示, 该方法的分类结果和病理学家的 诊 断 结 果 基 本 一 致 (非 加 权 Cohen ’s kappa 系 数 为 0.615); 该值高于最初的医院诊断( 0.524)。 Nitin 等[44] 提出了一种深度学习方法, 用于检测前列腺活检图的癌 症组织并预测其 GS。该方法是一种半监督方法, 采用 U 型架构 U-Net 并使用主动学习和不确定性措施来注释未 标注的样本。其在 425 个核心针活检 WSI 的内部测试集 上 准 确 率 为 89.4% , kquad(二 次 加 权 kappa) 为 0.92; 在 1 201 张 图 像 的 外 部 测 试 集 上 , 准 确 性 为 85.3%, kquad 为 0.96 。Valentin 等[45]提出了利用 SegGini 来评定前 列腺癌 GS,这是一种使用图神经网络的弱监督方法, 它 可以利用多重弱监督注释, 即不精确和不完整的注释来 分割活检病理图像。首先, SegGini 为输入的组织学图像 构建组织图表示; 然后, 通过使用不精确的标签、不完 整的标签或两者都使用来实现弱监督学习。
3 展望
深度学习技术自出现以来就深受欢迎, 其在自动学 习图像特征方面具有强大的能力, 近年来出现了一些经 典的模型, 如 VGG 、UNet 、DenseNet 等, 这些网络模型 改造后被运用于前列腺癌 Gleason 评分,并取得了优异的 成果, 但仍然有一些不足之处。由于不同医院之间的不 同仪器存在差异, 即使是相同类型的仪器之间也存在差 异, 因此要判断一个模型的效果只在一个数据集中验证 是不够的, 这些模型仍需要经过大量的外部验证。血液 中的微量元素能有效辅助诊断前列腺癌, 研究发现, 许 多肿瘤的发生、进展与微量元素的不足或过量密切相 关[5]。由微量元素引起的代谢变化应该考虑到前列腺癌 变的过程[6]。前列腺特异性抗原是前列腺癌的特异性标 志物, 结合 PSA 等的多模态信息能有效辅助诊断前列腺 癌。对于面向核磁共振前列腺癌 Gleason 评分的深度学习 方法而言, 纳入更多的核磁共振序列进行分析是未来该 领域发展的趋势, 因为不同序列的核磁共振包含不同的 信息, 有利于模型学习到更多的信息。通常, 在 MRI 中 会出现前列腺病灶目标较小, 在极其复杂的背景下难以 被发现的情况, 因此如何提高网络对于病变组织及其周 围非病变组织体素的判别力是未来该领域发展的一个方 向。在面向前列腺癌 Gleason 评分的深度学习方法研究中, 半监督的学习方法是热点, 使用半监督学习能够减 少大量的标注工作以及减少噪声标签的引入。
4 结束语
本文回顾了面向前列腺癌 Gleason 评分的深度学习方 法的发展, 以不同类型医学临床数据为研究对象, 面向 前列腺癌 Gleason 评分的深度学习方法可以分为面向核磁 共振图像、面向活检组织病理图前列腺癌 Gleason 评分的 深度学习方法。此类方法使用到经典的深度学习模型, 对于前列腺癌 Gleason 评分有优异的效果, 可以为前列腺 癌的临床诊断提供有意义的参考, 但其在大量外部验证 和进一步提高准确率方面仍有待提高。
参考文献:
[1] BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global can ‐ cer statistics 2018: GLOBOCAN estimates of incidence and mor ‐ tality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2018. 68(6): 394-424.
[2] SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality world ‐ wide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2021. 71(3): 209-249.
[3] ADEDIRAN A, OLATUNBOSUN E J G J O H S. A descriptive study of prostate lesions in largest hospital in ONDO state of Ni ‐ geria [J]. 2020. 5(2): 18-39.
[4] KANNA G P, KUMAR S J K J, PARTHASARATHI P, et al. A re ‐ view on prediction and prognosis of the prostate cancer and glea ‐ son grading of prostatic carcinoma using deep transfer learning based approaches[J]. Archives of Computational Methods in En ‐ gineering, 2023. 30(5): 3113-3132.
[5] 刘申 , 吴小候 . 前列腺癌诊断的研究新进展[J]. 重庆医学 , 2017. 46(15): 2150-2152.
[6] 邢朋毅, 孟英豪, 马超, 等 . MRI 纹理分析联合机器学习模型 对前列腺腺泡癌 Gleason 分级的预测价值[J]. 生物医学工程 与临床, 2022. 26(2): 174-180.
[7] FEHR D, VEERARAGHAVAN H, WIBMER A, et al. Automatic classification of prostate cancer Gleason scores from multipara ‐ metric magnetic resonance images[J]. Proceedings of the Nation ‐ al Academy of Sciences of the United States of America, 2015. 112(46): E6265-E6273.
[8] LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015. 521: 436-444.
[9] 李文星, 唐军, 屈艺, 等 . 人工智能在医学教育中的应用和发 展[J]. 成都中医药大学学报(教育科学版), 2019. 21(1): 17-18.
[10] 宋涛, 陆建平, 张倩雯 . 人工智能医学影像技术在胰腺神经 内分泌肿瘤分级中的应用[J]. 第二军医大学学报, 2020. 41 (4): 433-438.
[11] ESTEVA A, KUPREL B, NOVOA R A, et al. Dermatologist- level classification of skin cancer with deep neural networks[J]. Nature, 2017. 542: 115-118.
[12] RAJPURKAR P, IRVIN J, ZHU K, et al. CheXNet: radiologist- level pneumonia detection on chest X-rays with deep learning [J]. ArXiv e-Prints, 2017: arXiv: 1711.05225.
[13] POPLIN R, VARADARAJAN A V, BLUMER K, et al. Predic ‐ tion of cardiovascular risk factors from retinal fundus photo ‐ graphs via deep learning[J]. Nature Biomedical Engineering,2018. 2: 158-164.
[14] ANGERMUELLER C, PÄRNAMAA T, PARTS L, et al. Deep learning for computational biology[J]. Molecular Systems Biolo ‐ gy, 2016. 12(7): 878.
[15] GLEASON D F. Histologic grading of prostate cancer: a per‐ spective[J]. Human Pathology, 1992. 23(3): 273-279.
[16] OZKAN T A, ERUYAR A T, CEBECI O O, et al. Interobserver variability in Gleason histological grading of prostate cancer[J]. Scandinavian Journal of Urology, 2016. 50(6): 420-424.
[17] SAUTER G, STEURER S, CLAUDITZ T S, et al. Clinical utility of quantitative gleason grading in prostate biopsies and prosta ‐ tectomy specimens[J]. European Urology, 2016. 69(4): 592-598.
[18] KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[J]. Com ‐ munications of the ACM, 2017. 60(6): 84-90.
[19] DONATI O F, AFAQ A, VARGAS H A, et al. Prostate MRI: evaluating tumor volume and apparent diffusion coefficient as surrogate biomarkers for predicting tumor Gleason score[J]. Clinical Cancer Research: an Official Journal of the American Association for Cancer Research, 2014. 20(14): 3705-3711.
[20] WANG Q, LI H, YAN X, et al. Histogram analysis of diffusion kurtosis magnetic resonance imaging in differentiation of patho ‐ logic Gleason grade of prostate cancer[J]. Urologic Oncology: Seminars and Original Investigations, 2015. 33(8): 337. e15- 337.e24.
[21] OSMAN S O S, LEIJENAAR R T H, COLE A J, et al. Comput ‐ ed tomography-based radiomics for risk stratification in pros ‐ tate cancer[J]. International Journal of Radiation Oncology, Biol ‐ ogy, Physics, 2019. 105(2): 448-456.
[22] AHMED H U, BOSAILY A E S, BROWN L C, et al. Diagnostic accuracy of multi-parametric MRI and TRUS biopsy in prostate cancer (PROMIS): a paired validating confirmatory study[J]. Lancet, 2017. 389(10071): 815-822.
[23] CAO R M, MOHAMMADIAN BAJGIRAN A, AFSHARI MI ‐ RAK S, et al. Joint prostate cancer detection and gleason score prediction in mp-MRI via FocalNet[J]. IEEE Transactions on Medical Imaging, 2019. 38(11): 2496-2506.
[24] HE K M, GKIOXARI G, DOLLÁR P, et al. Mask R-CNN[C]// 2017 IEEE International Conference on Computer Vision (IC ‐ CV). Venice, Italy. IEEE, 2017: 2980-2988.
[25] JAEGER P F, KOHL S A A, BICKELHAUPT S, et al. Retina U-net: embarrassingly simple exploitation of segmentation su ‐ pervision for medical object detection[EB/OL]. 2018: arXiv: 1811.08661. http://arxiv.org/abs/1811.08661.pdf.
[26] PELLICER-VALERO O J, MARENCO JIMÉNEZ J L, GONZA ‐ LEZ-PEREZ V, et al. Deep learning for fully automatic detec ‐ tion, segmentation, and Gleason grade estimation of prostate cancer in multiparametric magnetic resonance images[J]. Scien ‐ tific Reports, 2022. 12: 2975.
[27] LIN T Y, GOYAL P, GIRSHICK R, et al. Focal loss for dense object detection[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020. 42(2): 318-327.
[28] VENTE C, VOS P, HOSSEINZADEH M, et al. Deep learning regression for prostate cancer detection and grading in Bi-para ‐ metric MRI[J]. IEEE Transactions on Bio-Medical Engineering, 2021. 68(2): 374-383.
[29] CHADDAD A, KUCHARCZYK M J, DESROSIERS C, et al.Deep radiomic analysis to predict gleason score in prostate can ‐cer[J]. IEEE Access, 2020. 8: 167767-167778.
[30] BRUNESE L, MERCALDO F, REGINELLI A, et al. Radiomics for gleason score detection through deep learning[J]. Sensors, 2020. 20(18): 5411.
[31] KÄLLÉN H, MOLIN J, HEYDEN A, et al. Towards grading gleason score using generically trained deep convolutional neu ‐ ral networks[C]//2016 IEEE 13th International Symposium on Biomedical Imaging (ISBI). Prague, Czech Republic. IEEE, 2016: 1163-1167.
[32] AZIZPOUR H, RAZAVIAN A S, SULLIVAN J, et al. Factors of transferability for a generic ConvNet representation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2016. 38(9): 1790-1802.
[33] SERMANET P, EIGEN D, ZHANG X, et al. OverFeat: integrat ‐ ed Recognition, Localization and Detection using Convolutional Networks[EB/OL]. 2013: arXiv: 1312.6229. http://arxiv.org/abs/ 1312.6229.pdf.
[34] BULTEN W, PINCKAERS H, VAN BOVEN H, et al. Automat ‐ ed Gleason Grading of Prostate Biopsies using Deep Learning [EB/OL]. 2019: arXiv: 1907.07980. http://arxiv. org/abs/ 1907.07980.pdf.
[35] LOKHANDE A, BONTHU S, SINGHAL N. Carcino-net: a deep learning framework for automated gleason grading of prostate bi ‐ opsies[C]//Proceedings of 2020 42nd Annual International Con ‐ ference of the IEEE Engineering in Medicine & Biology Society (EMBC). Canada: IEEE, 2020: 1380-1383.
[36] HE K M, ZHANG X Y, REN S Q, et al. Spatial pyramid pooling in deep convolutional networks for visual recognition[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2015. 37(9): 1904-1916.
[37] KOTT O, LINSLEY D, AMIN A, et al. Development of a deep learning algorithm for the histopathologic diagnosis and gleason grading of prostate cancer biopsies: a pilot study[J]. European Urology Focus, 2021. 7(2): 347-351.
[38] HE K, ZHANG X, REN S, et al. Identity mappings in deep re ‐ sidual networks[C]//Proceedings of the Computer Vision – EC ‐ CV 2016: 14th European Conference. Netherlands, 2016.
[39] KALAPAHAR A, SILVA-RODRÍGUEZ J, COLOMER A, et al.Gleason grading of histology prostate images through semantic segmentation via residual U-net[C]//Proceedings of 2020 IEEE International Conference on Image Processing (ICIP). Abu Dha ‐ bi:IEEE, 2020: 2501-2505.
[40] BADRINARAYANAN V, KENDALL A, CIPOLLA R. SegNet: a deep convolutional encoder-decoder architecture for image segmentation[J]. IEEE Transactions on Pattern Analysis and Ma ‐ chine Intelligence, 2017. 39(12): 2481-2495.
[41] RYU H S, JIN M S, PARK J H, et al. Automated gleason scor‐ ing and tumor quantification in prostate core needle biopsy im ‐ ages using deep neural networks and its comparison with pathol ‐ ogist-based assessment[J]. Cancers, 2019. 11(12): 1860.
[42] CHEN L C, ZHU Y K, PAPANDREOU G, et al. Encoder-de ‐ coder with atrous separable convolution for semantic image seg ‐ mentation[C]//Computer Vision – ECCV 2018: 15th European Conference, Munich, Germany, September 8 – 14. 2018. Pro ‐ ceedings, Part VII. ACM, 2018: 833 –851.
[43] WANG XL, GIRSHICK R, GUPTA A, et al. Non-local neural net ‐ works[C]//Proceedings of 2018 IEEE/CVF Conference on Com ‐ puter Vision and Pattern Recognition.IEEE, 2018: 7794-7803.
[44] SINGHAL N, SONI S, BONTHU S, et al. A deep learning system for prostate cancer diagnosis and grading in whole slide images of core needle biopsies[J]. Scientific Reports, 2022. 12: 3383.
[45] ANKLIN V, PATI P, JAUME G, et al. Learning whole-slideseg ‐ mentation from inexact and incomplete labels using tissue graphs[C]//Proceedings of International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2021: 636-646.










