群体感应介导的乳酸菌益生作用研究进展论文
2024-09-26 11:35:53 来源: 作者:dingchenxi
摘要:近年来,乳酸菌的益生特性及机制得到了广泛研究,但大多数研究集中于单菌的内在调控机制上。群体感应(Quorum sensing,QS)作为一种基于信号分子的细菌通信机制,在乳酸菌生长代谢和抵抗环境应激的过程中具有重要作用。
摘要:近年来,乳酸菌的益生特性及机制得到了广泛研究,但大多数研究集中于单菌的内在调控机制上。群体感应(Quorum sensing,QS)作为一种基于信号分子的细菌通信机制,在乳酸菌生长代谢和抵抗环境应激的过程中具有重要作用。本文介绍了乳酸菌中的3种QS类型及其作用机制,综述了QS调控乳酸菌生物膜形成、细菌素产生、耐酸特性等生理功能的作用机制,并阐述了QS介导乳酸菌益生特性和对致病菌拮抗作用的调控机制,为乳酸菌益生功能的研究与应用提供了理论指导。
关键词:乳酸菌;群体感应;益生作用
乳酸菌(Lactic acid bacteria,LAB)是一类通过发酵碳水化合物获得能量并主要产生乳酸的微生物,具有多种益生特性,如抗氧化活性,改善乳糖不耐、缓解腹泻、预防胃溃疡、提高宿主免疫力等[1-4]。深入了解乳酸菌发挥益生作用的分子机制有助于其在食品和医药领域的应用。
群体感应(Quorum sensing,QS)是一种基于信号分子的细胞间通讯,广泛存在于真菌、细菌甚至病毒中[5]。QS通过自动诱导剂(Autoinducer,AIs)这一信号分子的产生、释放和检测,获得临近群落细胞密度和物种组成的相关信息,从而调整其生理生化功能[6]。QS依赖细菌细胞密度调节基因表达,当信号分子积累达到阈值时,可以被细菌感知,从而在整个微生物群落中触发协同生理功能,包括生物发光、形成生物膜、表达毒力因子、合成抗生素和细菌素、产生代谢物等[7]。研究表明,乳酸菌具有合成细菌素[8]、形成生物膜[9]、耐酸耐胆盐[10]等多种与QS密切相关的生理特性。本文综述了乳酸菌中三种QS系统、QS系统是如何调控乳酸菌的生理功能和QS是如何影响乳酸菌的益生作用的。
1群体感应
1.1 QS分类与机制
根据信号分子的不同,QS系统主要分为四种类型:(1)LuxI/LuxR QS系统,主要存在于革兰氏阴性菌中。LuxI型蛋白通过自由扩散穿过细菌细胞膜,在胞外积累达到阈值浓度时,AHLs分子会重新进入细胞,LuxR样蛋白与这一同源自动诱导剂结合形成LuxR-AHLs复合物,随后与靶基因启动子结合并激活转录,调控基因表达[11]。(2)AIP/TCS QS系统,这是一种双组份系统。经典的TCS由跨膜受体组氨酸蛋白激酶(Histidine protein kinase,HPK)和同源细胞质反应调节子(Response regulator,RR)组成[12]。(3)AI-2/LuxS QS系统,介导细菌种内和种间通信。AI-2信号分子(Autoinducer-2,AI-2)由LuxS蛋白催化合成,luxS基因广泛存在于革兰氏阳性和阴性细菌中,具有较高的同源保守性[11]。(4)AI-3介导的QS系统。近年来,相继在某些细菌中发现了新的信号分子,如肠出血性大肠杆菌(Enterohemorrhagic E.coli,EHEC)的LuxS蛋白发生突变后不能合成AI-2,但可以产生另一自诱导物AI-3(Autoinducer-3,AI-3),并通过QS系统调节该菌的运动性、黏附性以及致病性[13]。
1.2乳酸菌中的QS系统
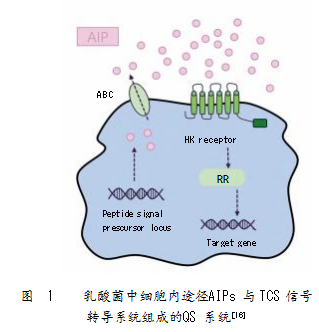
乳酸菌的QS系统大体分为种内信息交流和种间信息交流两种。种内信息交流的信号分子AI-1是一种小分子自身诱导信号肽AIP,也是寡肽切割修饰的产物[14],有两种不同的信号感知途径。
第一种为AIP/TCS途径(图1)。随着乳酸菌增殖,AIP可由前导肽经修饰产生,也可由脂蛋白水解产生[15]。当AIP在胞外累计达到阈值时,可被TCS的HPK识别,后者磷酸化自身保守的组氨酸残基,将磷酸化基团转移到RR的保守天冬氨酸残基上,以调控相关基因的表达[16]。
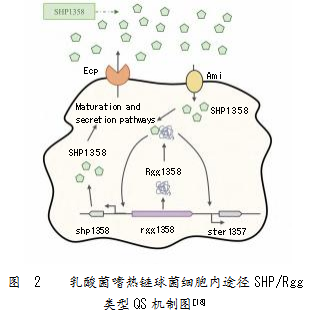
第二种为SHP/Rgg途径(Short hydrophobic peptide,SHP)。由ABC家族转运蛋白组成的寡肽Opp渗透酶转运系统将AIP重新运输至细胞内,AIP与转录调节因子或Rap蛋白相互作用,后两者都属于RNPP家族(包括Rap,Rgg,NprR,PlcR和PrgX),可以调节特定基因的表达[15]。嗜热链球菌(Streptococcus thermophilus LMD-9)中存在由短疏水肽SHP和Rgg家族的转录调节因子组成的SHP/Rgg QS系统(图2)[17]。SHP1358被跨膜内肽酶Eep切割转运至胞外,随着细菌细胞密度的增加,SHP累计到一定的阈值后,经Opp寡肽通透酶系统重新转运至胞内,作用于STER-1357基因的转调控蛋白STER-1358(Rgg)。STER-1357基因转录的产物进而调控下游基因表达,对外界环境作出应答[18]。
乳酸菌种间进行信息交流的信号分子为AI-2。AI-2/LuxS QS系统在细菌中普遍存在,通常与AHLs或AIP介导的QS系统共同发挥作用[19]。LuxS酶催化产生AI-2,在细胞群体密度高时,AI-2被受体蛋白LuxP检测到并与之结合,形成的AI-2/LuxP复合物与激活磷酸转移酶LuxU的LuxQ蛋白激酶相互作用,后者磷酸化LuxO转录调节因子,诱导LuxS转录[20,21]。这种机制在乳酸菌中广泛存在。
1.3不同乳酸菌的QS
乳酸菌具有不同的QS调节方式。发酵乳杆菌2-1中存在AI-2/LuxS QS系统,信号分子为AI-2[22]。鼠李糖乳杆菌GG、鼠李糖乳杆菌BFE5264中同样被证实具有AI-2/LuxS系统[23-24]。植物乳杆菌中发现的QS系统较多,其中植物乳杆菌NR74、299v中的AI-2/LuxS发挥作用[23],而植物乳杆菌WCFS1中被证实存在与金黄色葡萄球菌同源的lamBDCA系统[25],植物乳杆菌NC8中存在AIP/TCS系统[26]。嗜酸乳杆菌NCFM[27]、长双歧杆菌NCC2705[28]中同样检测到了AI-2/LuxS系统。嗜酸乳杆菌La-5可产生细菌素lactacin B,与TCS双组分系统关联形成QS系统[29]。嗜热链球菌LMD-9可由AIP/TCS系统调控产生二类细菌素[30],存在一种特殊的Rgg/SHP系统,由Rgg家族转录调控因子和信号分子SHP组成。
2 QS调控乳酸菌生理功能
2.1 QS系统调控乳酸菌形成生物膜
细菌生物膜指黏附于生命体或非生命体表面形成的一种高度组织化的复杂微生物菌落聚集体,被细菌生长繁殖过程中分泌的胞外基质包裹在内形成的膜状结构[31]。研究表明,生物膜的形成增强了乳酸菌的抗炎特性与免疫调节作用[32],使乳酸菌具有了更强大的耐环境胁迫和黏附于肠道内壁的能力[33]。
菌体的聚集积累与其黏附性密切相关,对生物膜的形成具有促进作用[34-35]。LuxS基因可以影响乳酸菌生物膜的形成情况。Gerald等研究发现罗伊氏乳杆菌100-23C LuxS基因缺失突变株形成的生物膜厚于野生型,且添加AI-2也无法复原[36]。在嗜柠檬酸明串珠菌37中的qPCR反应揭示了处于生物膜状态的细胞中LuxS和pfs基因的表达量相比浮游状细胞均有所上调,与AI-2的活性变化趋势一致,结合已有研究可得出结论,LuxS基因可能为调控生物膜形成的关键基因,pfs基因可能会参与调节生物膜形成[37]。Gu[9]等研究发现外源添加AI-2会使植物乳杆菌的lamC和ftsH基因表达受到抑制,但能促进生物膜和胞外多糖(Exopolysaccharide,EPS)的形成,说明lamC和ftsH基因可能是调控植物乳杆菌生物膜形成的关键基因。因此,推测乳酸菌生物膜的形成可能与AI-2无直接介导关系,但LuxS等关键基因的表达是不可或缺。
目前对AI-2/LuxS型QS中上游LuxS基因的调控机制较为成熟,而下游生物膜形成的具体路径则有待研究。
2.2 QS调节乳酸菌合成细菌素
细菌素作为一类由细菌核糖体合成的有抗菌活性的蛋白质或多肽,具备影响其他细菌生物膜形成、阻碍蛋白功能发挥等作用[38]。乳酸菌所产的细菌
素在体内可被蛋白酶消化,具有高效、无毒、耐酸、耐
高温、无残留、不产抗药性、抑制腐败和病原微生物繁殖的特点,在不损害食物安全性的情况下能够增强食品的感官吸引力和营养特性[39,40],因此作为一种天然的食品防腐剂得到了广泛应用。
已有大量研究表明QS系统能够调控乳酸菌细菌素合成。乳酸乳球菌产生的乳链菌肽(Nisin)是目前研究最透彻的I型细菌素,是由组氨酸蛋白激酶NisK和应答调控蛋白NisR组成的一种非典型性TCS介导。Nisin作为信号分子积累到一定阈值时可
以被TCS识别,表达合成细菌素的基因[41]。当NisK或NisR遭到破坏时,细菌不能合成Nisin。
乳酸菌合成的细菌素通常属于Ⅱ类细菌素。已经证实植物乳杆菌C11,WCFS1,NC8,J23等菌株具有相同或相似的QS系统,可以调控细菌素的合成。植物乳杆菌C11中调控细菌素生成的基因座由5个操纵子组成,分别为:plnABCD、plnEFI、plnJKLR、plnMNOP和plnGHSTUVW。操纵子plnABCD属于QS系统,受Plantaricin A基因编码的AIP诱导。植物乳杆菌WCFS1中的pln基因座与植物乳杆菌C11的结构和序列一致,植物乳杆菌J51与植物乳杆菌C11调控细菌素的相关QS基因一致,因此具有相同的QS调节细菌素合成机制[8,42]。植物乳杆菌NC8、J23含有与C11相似的合成细菌素基因位点,包含许多相似的操纵子[43]。但NC8菌株的AIP/TCS系统plnNC8IF-plnNC8HK-plnD这三个操纵子编码产生,其中plnc8IF编码AIP,plnc8HK基因编码产生HPK,与C11-plnD高度相似的NC8-plnD编码RR。嗜热链球菌LMD-9中的blp基因簇具有Ⅱ类细菌素基因座的所有特征,其中包含编码合成细菌素和QS系统的相关基因[30]。
目前,有关Ⅲ类细菌素的研究较少,且其是否与QS相关并未有明确报道。QS系统参与调控多种乳酸菌细菌素合成,但潜在机制尚未研究透彻,通过调节QS系统进而提高细菌素合成量是一个新的研究方向。
2.3 QS调控乳酸菌耐酸性
乳酸菌在肠道中定植存活,耐受胃肠道中的酸胁迫和高渗透压胁迫,必然具备一定的耐酸性。有研究表明,乳酸菌可以通过LuxS/AI-2 QS系统调控应答环境刺激,调节自身的生长代谢[23]。鼠李糖乳杆菌GG的LuxS突变体与野生型相比,在小鼠胃液中的存活率显著降低,表明QS系统与鼠李糖乳杆菌的胃酸应激与定植相关[24]。Gu等发现酸胁迫会对发酵乳杆菌2-1和粪肠球菌8-3产生影响,增加AI-2的活性,增加LuxS和pfs的基因表达量[22]。通过全基因组芯片技术分析,发现在酸性环境下,LuxS基因在嗜酸乳杆菌HPK突变株中表达量显著增加。尽管如此,突变株相较于野生型仍然对酸胁迫更加敏感[44]。这些研究表明酸胁迫环境下,乳酸菌可通过QS中的LuxS/AI-2系统,提高LuxS基因转录水平,增强AI-2信号分子的活性,进而增强其对酸的耐受性使其更适应酸性环境。
3乳酸菌益生作用与QS
3.1乳酸菌益生作用
乳酸菌作为一种重要的益生菌,具有调节肠道相关疾病、调节宿主免疫反应、缓解酒精性肝病和合成多种活性化合物等生理功能,在乳制品发酵、果蔬以及谷物加工和天然食品防腐剂等领域得到了广泛应用。
研究表明,乳酸菌能够调节肠道菌群和肠道免疫力、抑制致病酶的活性,保护宿主黏膜的屏障功能、维持肠上皮的再生和稳态,修复病理损伤造成的肠道损伤,改善结肠癌的症状[45]。近年来,大量研究表明QS系统参与乳酸菌益生作用的调控机制复杂(图3)。低pH或高浓度胆盐条件下,QS系统所介导分泌的AI-2/AIP可促进乳酸菌EPS产生[9]、生物膜形成[10]、增强其对环境的抵抗能力,通过QS系统的调控,乳酸菌得以合成细菌素[41],且更容易定植于肠道,发挥益生作用。在受到致病菌感染的体外和体内环境中,因乳酸菌的拮抗特性,致病菌形成生物膜[46]、产生毒力因子[47]等生理功能将受到抑制。

3.2.1 QS参与调控乳酸菌益生作用
乳酸菌具有多种与QS相关的发挥益生功能的途径。植物乳杆菌L3产生的苯乳酸(Phenyllactic acid,PLA)是一种具有抗菌活性的天然有机酸,作为新型防腐剂在食品工业得到了广泛应用。Chai等[49]发现LuxS蛋白是PLA合成过程中的关键蛋白,随着AI-2浓度的增加,植物乳杆菌L3的细胞密度和PLA产量呈增加趋势。当发酵液中QS系统受到抑制时,AI-2浓度、PLA产量和细胞密度降低,证实植物乳杆菌L3中的PLA合成由LuxS/AI-2系统调节。
许多乳酸菌的益生作用源于细菌素的产生。植物乳杆菌NC8[26]、乳酸乳球菌、嗜酸乳杆菌La-5[29]、植物乳杆菌DC400分别产生PLNC8IF、Nisin、lactacin B、PlnA细菌素作为AIP,与TCS共同调节细菌素的合成。通过构建植物乳杆菌KLDS1.0391菌株LuxS突变株,发现AI-2活性显著低于野生菌株,细菌素产量显著降低,改变了其生长特性与益生作用[50]。
乳酸菌生物膜抵抗环境应激的特性是其发挥益生作用的重要途径。大量研究表明,QS系统可以调节乳酸菌生物膜形成,改善其耐酸和耐胆盐性,提高其生存能力。Zhang等[10]研究发现在酸性环境、低胆盐浓度和胃液环境中生物膜对旧金山乳酸菌具有保护作用。外源添加AI-2可以显著促进菌株生长和生物膜形成,提高了胆盐耐受能力,可能由于黏度增加,与对照组相比,添加AI-2后生物膜表面更加光滑,细胞分布致密。当胆盐浓度过高时,旧金山乳酸菌存活状态与浮游态菌株没有差异性,表明生物膜的保护能力有限。
在细菌共培养条件下,QS系统调节的乳酸菌具有更高的生物膜形成和存活能力,可以更好地发挥其益生功能。Wen等[51]发现将嗜酸乳杆菌与双歧杆菌共培养,由于双歧杆菌具有产酸能力,环境pH会降低,能够促进嗜酸乳杆菌生长、QS介导的LuxS基因表达、AI-2释放。同时共培养条件显著促进了生物膜的产生,保护了嗜酸乳杆菌在胃肠道环境中的有效定植。植物乳杆菌DC400和旧金山乳杆菌DPPMA174或罗伊氏乳杆菌A7共培养时,LuxS基因的表达量为单独培养时的2.5倍和3.5倍,植物乳杆菌DC400其他QS相关基因/蛋白质的表达、AI-2类物质的合成也会受到影响。植物乳杆菌AB-1和干酪乳杆菌共培养时植物乳杆菌AB-1的抗菌活性显著增强,LuxS基因和细菌素调节操纵子plnB和plnC的转录水平显著上调,该研究表明LuxS/AI-2系统除了调节细菌素合成,也可能影响乳酸菌共培养的协同作用[52]。
以上研究表明乳酸菌益生作用的发挥与QS系统密切相关,具体体现在细菌素合成,生物膜形成,耐受强酸、高胆盐浓度等环境胁迫等方面。QS介导乳酸菌与其他菌株共培养的发酵过程,可以增加菌株的黏附能力,为细菌间相互交流与作用提供平台,能有效增强乳酸菌在胃肠道环境中的存活与定植能力。
3.2.2 QS参与调控乳酸菌拮抗致病菌
除了自身及其代谢产物具备益生功能,乳酸菌还可通过拮抗致病菌发挥益生作用,拮抗致病菌的途径中可能存在QS系统的调控作用。乳酸菌合成的细菌素AL705可以作为QS抑制剂,使单增李斯特菌信号分子失活,控制其生物膜的形成[53]。嗜酸乳杆菌NCFM与单增李斯特菌共培养,会显著抑制单增李斯特菌生长。嗜酸乳杆菌NCFM的LuxS基因受单增李斯特菌分泌物的影响显著上调,可能会调节嗜酸乳杆菌NCFM的生理行为,增强对单增李斯特菌的竞争性排斥,对肠道微生物群的细胞间通信产生积极影响[54]。
此外,乳酸菌可以通过干扰致病菌的QS对其产生拮抗作用。近期有研究表明昆基乳杆菌可以抑制铜绿假单胞菌生物膜的形成[46],一种由乳酸片球菌和植物乳杆菌产生的生物表面活性剂,能抑制金黄色葡萄球菌生物膜相关基因的表达,干扰其AI-2的释放和生物膜的形成,降低这一致病菌的黏附性[55]。与大肠杆菌共培养的鼠李糖乳杆菌GG微胶囊影响大肠杆菌LuxS相关基因的激活,持续抑制生物膜的形成。鼠李糖乳杆菌没有与大肠杆菌产生直接接触,大肠杆菌的QS受到抑制可能是由于AI-2等鼠李糖乳杆菌代谢产物的影响[56]。Wasfi等[57]研究发现乳酸菌能够使变形链球菌中QS基因VicKR和comCD的表达量下调,细胞黏附性降低,生物膜的形成与成熟受到阻碍,从而降低变形链球菌的毒性,抑制蛀牙和控制龋齿。植物乳杆菌ZX27上清液可以显著降低阴道加德纳氏菌中与毒力因子、生物膜形成、黏附力等相关基因的表达,干扰了其生物膜的形成[84]。综上所述,乳酸菌可以通过抑制病原微生物生长、生物膜形成及毒力因子表达达到抗菌效果,减少疾病的产生。一方面,乳酸菌自身QS系统发挥作用,拮抗致病菌;另一方面致病菌的QS系统也会受到乳酸菌抑制。
乳酸菌因具有益生作用受到了广泛研究,其调节肠道菌群紊乱、增强免疫力、保护肠道屏障功能等益生功能均与其黏附并定植于宿主肠道中息息相关。细菌素合成、生物膜形成、耐酸、耐胆盐等能力是乳酸菌益生作用发挥的前提,而这些生理功能皆受QS系统调控。此外,乳酸菌还可通过QS拮抗致病菌。目前,有关QS调控乳酸菌发挥益生功能的研究,多集中于LuxS/AI-2系统、生物膜、细菌素等经典研究方向,具体机制并未进行深入研究,对于QS系统调控下游基因的具体通路涉及较少,研究潜力巨大。
4结论与讨论
QS是乳酸菌感知外界环境变化并调节自身生理功能的重要信号转导机制,现有研究较少运用QS思路探究乳酸菌的益生作用机制,而多集中于乳酸菌本身。本文综述了乳酸菌中的QS系统、受QS调控的生理功能、QS参与的益生功能,具体内容为AIP/TCS、LuxS/AI-2和SHP/Rgg三种QS系统的作用机制,耐酸、耐胆盐、生物膜形成、细菌素产生、拮抗致病菌等重要生理功能作为乳酸菌发挥益生功能必不可少的前提与QS的关联。
鉴于现有研究成果,有关乳酸菌益生功能的研究大多集于乳酸菌自身和其代谢产物,与QS相关的机制研究较少,并不确定乳酸菌产生的益生特性是否与QS相关,因此无法通过调控QS系统进一步增强其益生特性。且在乳酸菌QS系统的研究中,有关LuxS/AI-2、AIP/TCS系统的探究较为普遍,内容多为QS中上游调控AI-2活性、LuxS基因和TCS相关基因的机制,而下游生物膜形成、细菌素合成、对酸和胆盐的耐受能力等生理功能的具体机制仍有待挖掘。为此,解析QS系统精准调控下游生理功能的机制有望成为乳酸菌研究领域的重要研究方向,为乳酸菌在高产细菌素、抗菌剂、食品防腐剂、膳食补充剂等领域的发展与应用提供理论指导。
参考文献
[1]GARBACZ K.Anticancer activity of lactic acid bacteria[J].Seminars in Cancer Biology,2022,86:356-66.
[2]MASOOD M I,QADIR M I,SHIRAZI J H,et al.Beneficial effects of lactic acid bacteria on human beings[J].Critical Reviews in Microbiology,2010,37(1):91-8.
[3]ZHANG Z W,LV J L,PAN L,et al.Roles and applications of probiotic Lactobacillus strains[J].Appl Microbiol Biot,2018,102(19):8135-43.
[4]VOUGIOUKLAKI D,TSIRONI T,TSANTES A G,et al.ProbioticProperties and Antioxidant Activity In Vitro ofLactic Acid Bacteria[J].Microorganisms,2023,11(5):1264.
[5]WU S B,LIU J H,LIU C J,et al.Quorum sensing for population-level control of bacteria and potential therapeutic applications[J].Cellular and Molecular Life Sciences,2020,77(7):1319-43.
[6]PAPENFORT K B B L.Quorum sensing signal response systems in Gram-negative bacteria[J].Nature Reviews Microbiology,2016,14(7):576-88.
[7]KAREB O,AIDER M.Quorum Sensing Circuits in the Communicating Mechanisms of Bacteria and Its Implication in the Biosynthesis of Bacteriocins by Lactic Acid Bacteria:a Review[J].Probiotics and Antimicrobial Proteins,2020,12(1):5-17.
[8]ROJO-BEZARES B,S iENZ Y,NAVARRO L,et al.Characterization of a new organization of the plantaricin locus in the inducible bacteriocin-producing Lactobacillus plantarum J23 of grape must origin[J].Archives of Microbiology,2008,189(5):491-9.
[9]GU Y,TIAN J,ZHANG Y,et al.Dissecting signal molecule AI-2 mediated biofilm formation and environmental tolerance in Lactobacillus plantarum[J].Journal of Bioscience and Bioengineering,2021,131(2):153-60.
[10]ZHANG Y,GU Y,WU R,et al.Exploring the relationship between the signal molecule AI-2 and the biofilm formation of Lactobacillus sanfranciscensis[J].LWT,2022,154:112704.
[11]CAGNO R D,ANGELIS M D,CALASSO M,et al.Proteomics of the bacterial cross-talk by quorum sensing[J].Journal of Biology,2011,74(1):19-34.
[12]SINGH A R.Regulation of Streptomyces Chitinases by Two-Component Signal Transduction Systems and their Post Translational Modifications:A Review[J].Journal of Pure and Applied Microbiology,2018,12(3):1417-33.
[13]WALTERS M,SIRCILI M P,SPERANDIO V.AI-3 Synthesis Is Not Dependent on luxS in Escherichia coli[J].Journal of Bacteriology,2006,188(16):5668-81.
[14]QIAN X,TIAN P,ZHAO J,et al.Quorum Sensing of Lactic Acid Bacteria:Progress and Insights[J].Food Reviews International,2022:1-12.
[15]MONNET V,JUILLARD V,GARDAN R.Peptide conversations inGram-positive bacteria[J].Critical Reviews in Microbiology,2016,42(3):339-51.
[16]WATERS C M,BASSLER B L.QUORUM SENSING:Cell-to-Cell Communication in Bacteria[J].Annual Review of Cell and Developmental Biology,2005,21(1):319-46.
[17]IBRAHIM M,GUILLOT A,WESSNER F,et al.Control of the Transcription of a Short Gene Encoding a Cyclic Peptide in Streptococcus thermophilus:a New Quorum-Sensing System?[J].Journal of Bacteriology,2007,189(24):8844-54.
[18]FLEUCHOT B,GITTON C,GUILLOT A,et al.Rgg proteins associated with internalized small hydrophobic peptides:a new quorum-sensing mechanism in streptococci[J].Molecular Microbiology,2011,80(4):1102-19.
[19]SELA S,FRANK S,BELAUSOV E,et al.A Mutation in the luxS Gene Influences Listeria monocytogenes Biofilm Formation[J].Applied and Environmental Microbiology,2006,72(8):5653-8.
[20]JOHANSEN P,JESPERSEN L.Impact of quorum sensing on the quality of fermented foods[J].Current Opinion in Food Science,2017,13:16-25.
[21]MARTINELLI D,BUNK M,CADALBERT B.Language of bacteria[J].Wochenblatt für Papierfabrikation,2002,130(14):973-5.
[22]GU Y,LI B,TIAN J,et al.The response of LuxS/AI-2 quorum sensing in Lactobacillus fermentum 2-1 to changes in environmental growth conditions[J].Annals of Microbiology,2018,68(5):287-94.
[23]YEO S,PARK H,JIY,et al.Influence ofgastrointestinal stress on autoinducer-2 activity of two Lactobacillus species[J].FEMS Microbiology Ecology,2015,91(7):fiv065.
[24]LEBEER S,CLAES INGMAR J J,VERHOEVEN TINE L A,et al.Impact of luxS and Suppressor Mutations on the Gastrointestinal Transit of Lactobacillus rhamnosus GG[J].Applied and Environmental Microbiology,2008,74(15):4711-8.
[25]STURME M H J,NAKAYAMA J,MOLENAAR D,et al.An agr-like two-component regulatory system in Lactobacillus plantarum is involved in production of a
novel cyclic peptide and regulation of adherence[J].Journal of Bacteriology,2005,187(15):5224-35.
[26]RUIZ-BARBA J L,CABALLERO-GUERRERO B,MALDONADO-BARRAGáN A,et al.Coculture with specific bacteria enhances survival of Lactobacillus plantarum NC8,an autoinducer-regulated bacteriocin producer,in olive fermentations[J].Food Microbiology,2010,27(3):413-7.
[27]BUCK BL,AZCARATE-PERIL MA,KLAENHAMMER T R.Role of autoinducer-2 on the adhesion ability of Lactobacillus acidophilus[J].J Appl Microbiol,2009,107(1):269-79.
[28]SUN Z,HE X,BRANCACCIO V F,et al.Bifidobacteria Exhibit LuxS-Dependent Autoinducer 2 Activity and Biofilm Formation[J].PLOS ONE,2014,9(2):e88260.
[29]TABASCO R,GARCíA-CAYUELA T,PELáEZ C,etal.Lactobacillus acidophilus La-5 increases lactacin B production when it senses live target bacteria[J].International Journal of Food Microbiology,2009,132(2):109-16.
[30]FONTAINE L,BOUTRY C,GUéDON E,et al.Quorum-Sensing Regulation of the Production of Blp Bacteriocins in Streptococcus thermophilus[J].Journal of Bacteriology,2007,189(20):7195-205.
[31]SOTO S M.Role of efflux pumps in the antibiotic resistance of bacteria embedded in a biofilm[J].Virulence,2013,4(3):223-9。
[32]RIEU A,AOUDIA N,JEGO G,et al.The biofilm mode of life boosts the anti-inflammatory properties of Lactobacillus[J].Cellular Microbiology,2014,16(12):1836-53.
[33]CHEOW W S,KIEW T Y,HADINOTO K.Controlled release of Lactobacillus rhamnosus biofilm probiotics from alginate-locust bean gum microcapsules[J].Carbohydrate Polymers,2014,103:587-95.
[34]LEBEER S,VERHOEVEN TINE L A,PEREA VéLEZ M,et al.Impact of Environmental and Genetic Factors on Biofilm Formation by the Probiotic Strain Lactobacillus rhamnosus GG[J].Applied andEnvironmentalMicrobiology,2007,73(21):6768-75.
[35]LEBEER S,VERHOEVEN TINE L A,FRANCIUS G,et al.Identification of a Gene Cluster for the Biosynthesis of a Long,Galactose-Rich Exopolysaccharide in Lactobacillus rhamnosus GG and Functional Analysis of the Priming Glycosyltransferase[J].Applied and Environmental Microbiology,2009,75(11):3554-63.
[36]TANNOCK GERALD W,GHAZALLY S,WALTER J,et al.Ecological Behavior of Lactobacillus reuteri 100-23 Is Affected by Mutation of the luxS Gene[J].Applied and Environmental Microbiology,2005,71(12):8419-25.
[37]YANG Q,WANG Y,AN Q,et al.Research on the role of LuxS/AI-2 quorum sensing in biofilm of Leuconostoccitreum 37 based on complete genome sequencing[J].3 Biotech,2021,11(4):189.
[38]CHOPRA L,SINGH G,KUMAR JENA K,et al.Sonorensin:A new bacteriocin with potential of ananti-biofilmagent and a food biopreservative[J].SciRep-Uk,2015,5(1):13412.
[39]NES I F,JOHNSBORG O.Exploration of antimicrobial potential in LAB by genomics[J].Current Opinion in Biotechnology,2004,15(2):100-4.
[40]CALO-MATA P,ARLINDO S,BOEHME K,et al.Current Applications and Future Trends of Lactic Acid Bacteria and their Bacteriocins for the Biopreservation of Aquatic Food Products[J].Food and Bioprocess Technology,2008,1(1):43-63.
[41]PATTON G C,VAN DER DONK W A.New developments in lantibiotic biosynthesis and mode ofaction[J].Current Opinion in Microbiology,2005,8(5):543-51.
[42]NAVARRO L,ROJO-BEZARES B,SáENZ Y,et al.Comparative study of the pln locus of the quorum-sensing regulated bacteriocin-producing L.plantarum J51 strain[J].International Journal of Food Microbiology,2008,128(2):390-4.
[43]STRAUME D,JOHANSEN R F,BJ?R?S M,et al.DNA binding kinetics of two response regulators,PlnC and PlnD,from the bacteriocin regulon of Lactobacillus plantarum C11[J].BMC Biochemistry,2009,10(1):17.
[44]AZCARATE-PERIL M A,MCAULIFFE O,ALTERMANNE,et al.Microarray Analysis of a Two-Component Regulatory System Involved in Acid Resistance and Proteolytic Activity in Lactobacillus acidophilus[J].Applied and Environmental Microbiology,2005,71(10):5794-804.
[45]CHEN Y,YANG B,ZHAO J,et al.Exploiting lactic acid bacteria for colorectal cancer:a recent update[J].Critical Reviews in Food Science and Nutrition,2022:1-17.
[46]BERRíOS P,FUENTES J A,SALAS D,et al.Inhibitory effect of biofilm-forming Lactobacillus kunkeei strains against virulent Pseudomonas aeruginosa in vitro and in honeycomb moth(Galleria mellonella)infection model[J].Beneficial Microbes,2018,9(2):257-68.
[47]QIAN Z,ZHU H,ZHAO D,et al.Probiotic Lactobacillus sp.Strains Inhibit Growth,Adhesion,Biofilm Formation,and Gene Expression of Bacterial Vaginosis-Inducing Gardnerella vaginalis[J/OL]2021,9(4):
[48]MOSLEHI-JENABIAN S,GORI K,JESPERSEN L.AI-2 signalling is induced by acidic shock in probiotic strains of Lactobacillus spp[J].International Journal of Food Microbiology,2009,135(3):295-302.
[49]CHAI Y,MA Q,NONG X,et al.Dissecting LuxS/AI-2 quorum sensing system-mediated phenyllactic acid production mechanisms of Lactiplantibacillus plantarum L3[J].Food Research International,2023,166:112582.
[50]JIA F-F,PANG X-H,ZHU D-Q,et al.Role of the luxS gene in bacteriocin biosynthesis by Lactobacillus plantarum KLDS1.0391:A proteomic analysis[J].Sci Rep-Uk,2017,7(1):13871.
[51]DI CAGNO R,DE ANGELIS M,CODA R,et al.Molecular adaptation of sourdough Lactobacillus plantarum DC400 under co-cultivation with other lactobacilli[J].Research in Microbiology,2009,160(5):358-66.
[52]LI J,YANG X,SHI G,et al.Cooperation of lactic acid bacteria regulated by the AI-2/LuxS system involve in the biopreservation of refrigerated shrimp[J].Food Research International,2019,120:679-87.
[53]MELIAN C,SEGLI F,GONZALEZ R,et al.Lactocin AL705 as quorum sensing inhibitor to control Listeria monocytogenes biofilm formation[J].JAppl Microbiol,2019,127(3):911-20.
[54]MOSLEHI-JENABIAN S,VOGENSEN FK,JESPERSEN L.The quorum sensing luxS gene is induced in Lactobacillus acidophilus NCFM in response to Listeria monocytogenes[J].International Journal of Food Microbiology,2011,149(3):269-73.
[55]YAN X,GU S,CUI X,et al.Antimicrobial,anti-adhesive and anti-biofilm potential of biosurfactants isolated from Pediococcus acidilactici and Lactobacillus plantarum against Staphylococcus aureus CMCC26003[J].Microbial Pathogenesis,2019,127:12-20.
[56]SONG H,ZHANG J,QU J,et al.Lactobacillus rhamnosus GG microcapsules inhibit Escherichia coli biofilm formation in coculture[J].Biotechnology Letters,2019,41(8):1007-14.
[57]WASFI R,ABD EL-RAHMAN O A,ZAFER M M,et al.Probiotic Lactobacillus sp.inhibit growth,biofilm formation and gene expression of caries-inducing Streptococcus mutans[J].Journal of Cellular and Molecular Medicine,2018,22(3):1972-83.










